 T-Gedächtniszelle nennt man eine Gedächtniszelle des Immunsystems, wenn sie sich aus einem aktivierten T-Lymphozyten entwickelt hat.
T-Gedächtniszelle nennt man eine Gedächtniszelle des Immunsystems, wenn sie sich aus einem aktivierten T-Lymphozyten entwickelt hat.Erklärungen für Wörter mit dem Anfangsbuchstaben T in meinen und den Hypertexten von Dr. Ingrid Schütt-Abraham
Roland Heynkes 3.9.2025, CC BY-SA-4.0 DE
If you cannot read German-language texts, you could try a translation into your native language with DeepL.
Navigiere mit den folgenden Links:
A B C D E F G H I J K L M N O P Q R S T U V W X Y Z
 T-Gedächtniszelle nennt man eine Gedächtniszelle des Immunsystems, wenn sie sich aus einem aktivierten T-Lymphozyten entwickelt hat.
T-Gedächtniszelle nennt man eine Gedächtniszelle des Immunsystems, wenn sie sich aus einem aktivierten T-Lymphozyten entwickelt hat.
 T-Helferzelle nennen Biologen T-Zellen, die mit ihren T-Zell-Rezeptoren spezifisch dem eigenen Körper fremde Antigene auf den MHC-2-Präsentiertellern Antigen präsentierender Zellen erkennen, dadurch aktiviert werden, sich durch Zellteilungen vermehren und dann andere weiße Blutkörperchen zum Kampf gegen Viren oder andere Mikroorganismen aktivieren. T-Helferzellen entstehen durch die T-Lymphopoese.
T-Helferzelle nennen Biologen T-Zellen, die mit ihren T-Zell-Rezeptoren spezifisch dem eigenen Körper fremde Antigene auf den MHC-2-Präsentiertellern Antigen präsentierender Zellen erkennen, dadurch aktiviert werden, sich durch Zellteilungen vermehren und dann andere weiße Blutkörperchen zum Kampf gegen Viren oder andere Mikroorganismen aktivieren. T-Helferzellen entstehen durch die T-Lymphopoese.
 T-Killerzelle, cytotoxische T-Zelle oder cytotoxischen T-Lymphozyt nennen Biologen T-Zellen, die mit ihren T-Zell-Rezeptoren spezifisch dem eigenen Körper fremde Antigene auf den MHC-1-Präsentiertellern anderer Körperzellen erkennen und diese daraufhin zum Selbstmord auffordern, um eine Krebserkrankung oder Virus-Infektion zu bekämpfen. Cytotoxische T-Lymphozyten entstehen durch die T-Lymphopoese.
T-Killerzelle, cytotoxische T-Zelle oder cytotoxischen T-Lymphozyt nennen Biologen T-Zellen, die mit ihren T-Zell-Rezeptoren spezifisch dem eigenen Körper fremde Antigene auf den MHC-1-Präsentiertellern anderer Körperzellen erkennen und diese daraufhin zum Selbstmord auffordern, um eine Krebserkrankung oder Virus-Infektion zu bekämpfen. Cytotoxische T-Lymphozyten entstehen durch die T-Lymphopoese.
 T-Lymphopoese (englisch: T lymphopoiesis) oder genauer T-Lymphozytopoese (englisch: T lymphocytopoiesis) nennt man die hauptsächlich im Knochenmark stattfindende Entwicklung zahlreicher T-Lymphozyten aus wenigen multipotenten (Manche nennen sie auch pluripotent.) hämopoetischen Stammzellen (HSC). Die Stammzellen können nur in bestimmten Nischen Stammzellen bleiben und nur eine von zwei Tochterzellen findet darin Platz. Der anderen Tochterzelle droht der Verlust der Unsterblichkeit, wenn sie keine andere freie Nische findet. So bleibt die Zahl der Stammzellen nahezu konstant. Nach dem Verlust der Stammzellfähigkeit nennt man die Nachkommen der zweiten Stammzell-Tochterzellen multipotent progenitors (MPP) oder multipotente Vorläuferzellen.
T-Lymphopoese (englisch: T lymphopoiesis) oder genauer T-Lymphozytopoese (englisch: T lymphocytopoiesis) nennt man die hauptsächlich im Knochenmark stattfindende Entwicklung zahlreicher T-Lymphozyten aus wenigen multipotenten (Manche nennen sie auch pluripotent.) hämopoetischen Stammzellen (HSC). Die Stammzellen können nur in bestimmten Nischen Stammzellen bleiben und nur eine von zwei Tochterzellen findet darin Platz. Der anderen Tochterzelle droht der Verlust der Unsterblichkeit, wenn sie keine andere freie Nische findet. So bleibt die Zahl der Stammzellen nahezu konstant. Nach dem Verlust der Stammzellfähigkeit nennt man die Nachkommen der zweiten Stammzell-Tochterzellen multipotent progenitors (MPP) oder multipotente Vorläuferzellen.
Wie fast immer in der Biologie ist die Sache natürlich auch in diesem Fall in Wirklichkeit sehr viel komplizierter. Unter anderem werden immer neue Untergruppen (Subtypen) von hämopoetischen Stammzellen (HSC) entdeckt. So wird inzwischen zwischen Langzeit- und Kurzzeit-Stammzellen unterschieden. Und man konnte nachweisen, dass es Stammzellen gibt, in denen bereits einzelne von den Genen aktiv sind, die eigentlich typisch für bestimmte spezialisiertere Zelltypen sind. Das bedeutet auch, dass aus der zweiten Tochterzelle der ursprünglichen Stammzelle nicht unmittelbar und nicht nur ein einziger multipotenter Progenitor (MPP) entsteht, sondern durch mehrere Zellteilungen entstehen mehrere MPP. Die Notwendigkeit der flexiblen Regulierbarkeit der Hämopoese spricht außerdem dafür, dass die Zahl der Zellteilungen zwischen der Langzeit-hämopoetischen Stammzelle und dem multipotenten Progenitor nach Bedarf flexibel eingestellt werden kann.
Wenn nötig, sind die MPP noch in der Lage, ihre Differenzierung zugunsten einer stärkeren Vermehrung zu verlangsamen. Die multipotenten Vorläuferzellen (MPP) teilen sich und ihre Tochterzellen verlieren langsam und anfangs noch reversibel einen Teil ihrer Multipotenz. Das bedeutet, dass sie sich für eine von mehreren möglichen Entwicklungslinien entscheiden. Im Falle der T-Lymphopoese entscheiden sie sich vorläufig gegen den myeloiden und für den lymphoiden Entwicklungsweg. Man bezeichnet diese noch reversibel auf Lymphopoese festgelegte Tochterzellen als lymphoid-primed multipotent / early lymphoid progenitors (LMPP/ELP).
Die LMPP/ELP teilen sich weiter und differenzieren sich weiter zu bei Mäusen common lymphoid progenitors (CLP) und bei Menschen Multi-lymphoid Progenitor (MLP) genannten Vorläuferzellen. Sie haben sich nun endgültig entschieden, B-Zellen, T-Zellen oder natürliche Killerzellen zu werden. Die CLP/MLP können sich auch nicht mehr selbst erneuern (ihre Differenzierung aufhalten).
Aber auch die CLP/MLP teilen und differenzieren sich weiter und ein Teil von ihnen verläßt das Knochenmark in Richtung Thymus. Dort reifen sie im Verlauf der Thymopoese zu T-Lymphozyten heran, während sie durch von außen nach innen durch Cortex und Medulla des Thymus wandern. Zu dieser Reifung gehört vor allem das individuelle (quasizufällige) Zusammensetzen einzigartiger Gene für die beiden T-Zell-Rezeptor-Ketten aus zahlreichen zur Auswahl stehenden Gen-Puzzleteilen. Und vom Ergebnis dieser T-Zell-Rezeptor-Reifung hängt das Schicksal des fertigen T-Lymphozyten ab.
Passt der T-Zell-Rezeptor zu keiner der beiden MHC-Präsentiertellersorten, dann ist die T-Zelle unbrauchbar und wird im Thymus getötet. Passt der T-Zell-Rezeptor zu MHC-1-Präsentiertellern, dann ist die T-Zelle ein cytotoxischer T-Lymphozyt (CTL oder T-Killerzelle). Passt der T-Zell-Rezeptor zu MHC-2-Präsentiertellern, dann ist der T-Lymphozyt eine T-Helferzelle. In beiden Fällen müssen die reifen T-Zellen aber noch die wichtigere Prüfung überstehen. T-Lymphozyten müssen nämlich unbedingt auch dann sterben, wenn ihre T-Zell-Rezeptoren körpereigene Peptide erkennen. Ansonsten könnte es zu einer Autoimmunkrankheit kommen.
Sind beide Prüfungen bestanden, dann verlassen die T-Lymphozyten den Thymus und wandern durch Blut, das Lymphsystem und andere Organe, wo sie bei Bedarf aktiviert werden.
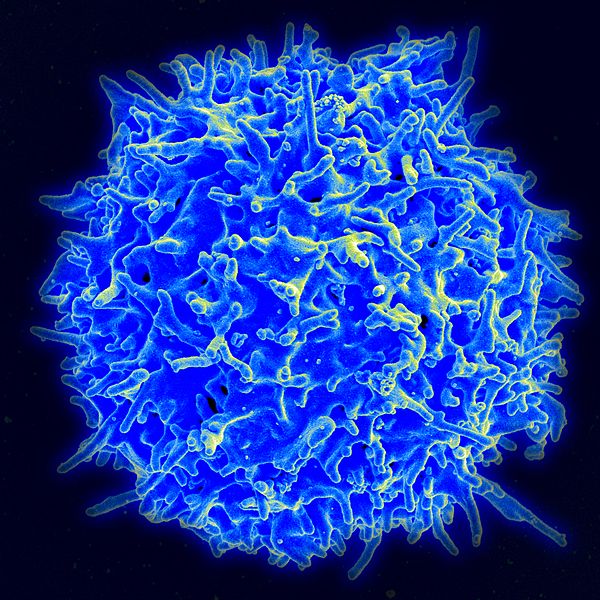
| T-Lymphozyt ist der wissenschaftliche Name für den gebräuchlicheren Begriff T-Zelle. T-Lymphozyten gehören zu den B-Lymphozyten, die wiederum zu den Leukozyten (weißen Blutkörperchen) gehören. |
 |
| National Institute of Allergy and Infectious Diseases (NIAID) im Verbund der National Institutes of Health (NIH), public domain |
 T-Zelle oder T-Lymphozyt nennen Biologen weiße Blutkörperchen (Leukozyt), die im Thymus oder einem Ersatzorgan einen besonderen Reifungs- und Selektionsprozess überlebt haben. Kommt ein noch unreifer Lymphozyt in den Thymus, dann reift er zur T-Zelle heran, indem er nach dem Zufallsprinzip einen einzigartigen Rezeptor bildet - den sogenannten T-Zell-Rezeptor. Sobald fertige T-Zell-Rezeptoren auf der Oberfläche einer neuen T-Zelle erscheinen, wird die T-Zelle zwei Tests unterzogen. Sie wird in den Selbstmord getrieben, wenn sie weder MHC-1- noch MHC-2-Präsentierteller erkennt. Und sie muss sterben, wenn sie Antigene des eigenen Körpers erkennt. Könnte eine T-Zelle nicht an Präsentierteller binden, dann wäre sie nur nutzlos. Erkennt aber eine T-Zelle ein Antigen des eigenen Körpers, dann kann sie eine kaum heilbare Autoimmunkrankheit verursachen und ist deshalb sehr gefährlich. Kann der T-Zell-Rezeptor einer T-Zelle an MHC-1-Präsentierteller ankoppeln und darauf nach Antigenen suchen, dann handelt es sich um eine T-Killerzelle. Erkennt ein T-Lymphozyt stattdessen MHC-2-Präsentierteller, dann nennt man ihn T-Helferzelle.
T-Zelle oder T-Lymphozyt nennen Biologen weiße Blutkörperchen (Leukozyt), die im Thymus oder einem Ersatzorgan einen besonderen Reifungs- und Selektionsprozess überlebt haben. Kommt ein noch unreifer Lymphozyt in den Thymus, dann reift er zur T-Zelle heran, indem er nach dem Zufallsprinzip einen einzigartigen Rezeptor bildet - den sogenannten T-Zell-Rezeptor. Sobald fertige T-Zell-Rezeptoren auf der Oberfläche einer neuen T-Zelle erscheinen, wird die T-Zelle zwei Tests unterzogen. Sie wird in den Selbstmord getrieben, wenn sie weder MHC-1- noch MHC-2-Präsentierteller erkennt. Und sie muss sterben, wenn sie Antigene des eigenen Körpers erkennt. Könnte eine T-Zelle nicht an Präsentierteller binden, dann wäre sie nur nutzlos. Erkennt aber eine T-Zelle ein Antigen des eigenen Körpers, dann kann sie eine kaum heilbare Autoimmunkrankheit verursachen und ist deshalb sehr gefährlich. Kann der T-Zell-Rezeptor einer T-Zelle an MHC-1-Präsentierteller ankoppeln und darauf nach Antigenen suchen, dann handelt es sich um eine T-Killerzelle. Erkennt ein T-Lymphozyt stattdessen MHC-2-Präsentierteller, dann nennt man ihn T-Helferzelle.
 T-Zell-Rezeptoren (TZR) ähneln Antikörpern, bleiben aber auf den Oberflächen von T-Zellen und erkennen spezifisch Antigene.
T-Zell-Rezeptoren (TZR) ähneln Antikörpern, bleiben aber auf den Oberflächen von T-Zellen und erkennen spezifisch Antigene.
 Taiga ist das russische Wort für den bis zur baumlosen Tundra reichenden nördlichen (borealen) Nadelwald. Es bedeutet: "dichter, undurchdringlicher, oft sumpfiger Wald". Wahrscheinlich haben Russen das Wort von Mongolen übernommen, die so ihren Bergwald nannten. Inzwischen wird der Begriff Taiga generell für alle nördlichen Nadelwälder verwendet. Typisch für alle borealen Wälder sind extrem kalte und lang andauernde Winter und relativ seltene Niederschläge. Für Laubbäume ist die Taiga aufgrund ihrer großen Blattoberflächen zu kalt und zu trocken.
Taiga ist das russische Wort für den bis zur baumlosen Tundra reichenden nördlichen (borealen) Nadelwald. Es bedeutet: "dichter, undurchdringlicher, oft sumpfiger Wald". Wahrscheinlich haben Russen das Wort von Mongolen übernommen, die so ihren Bergwald nannten. Inzwischen wird der Begriff Taiga generell für alle nördlichen Nadelwälder verwendet. Typisch für alle borealen Wälder sind extrem kalte und lang andauernde Winter und relativ seltene Niederschläge. Für Laubbäume ist die Taiga aufgrund ihrer großen Blattoberflächen zu kalt und zu trocken.
 Talent oder Begabung nennt man eine angeborene Fähigkeit, die bei entsprechendem Training zu überdurchschnittlichen Leistungen befähigt.
Talent oder Begabung nennt man eine angeborene Fähigkeit, die bei entsprechendem Training zu überdurchschnittlichen Leistungen befähigt.
 Talg nennt man einerseits das aus geschlachteten Wiederkäuern gewonnene feste Körperfett. Talg oder Hauttalg heißt aber auch das fettige Sekret der Talgdrüse, das unsere Haare sowie die Hornschicht der Oberhaut von außen einfettet und geschmeidig hält.
Talg nennt man einerseits das aus geschlachteten Wiederkäuern gewonnene feste Körperfett. Talg oder Hauttalg heißt aber auch das fettige Sekret der Talgdrüse, das unsere Haare sowie die Hornschicht der Oberhaut von außen einfettet und geschmeidig hält.
 Talgdrüsen sind holokrine Drüsen im oberen Teil der Lederhaut. Sie produzieren ein fettiges, Hauttalg genanntes Sekret, das unsere Haare sowie die Hornschicht der Oberhaut von außen einfettet und geschmeidig hält. Von den wenigen unbehaarten Hautbereichen und bestimmten Schleimhäuten abgesehen, findet man die Talgdrüsen am Epithelium der Haaranlagen. Daher nennt man sie auch Haarbalgdrüsen. Sie geben den Talg einfach auf die wachsenden Haare ab und an diesen entlang wandert der Talg auf die Haut. An Fußsohlen und Handinnenflächen besitzen wir keine Talgdrüsen.
Talgdrüsen sind holokrine Drüsen im oberen Teil der Lederhaut. Sie produzieren ein fettiges, Hauttalg genanntes Sekret, das unsere Haare sowie die Hornschicht der Oberhaut von außen einfettet und geschmeidig hält. Von den wenigen unbehaarten Hautbereichen und bestimmten Schleimhäuten abgesehen, findet man die Talgdrüsen am Epithelium der Haaranlagen. Daher nennt man sie auch Haarbalgdrüsen. Sie geben den Talg einfach auf die wachsenden Haare ab und an diesen entlang wandert der Talg auf die Haut. An Fußsohlen und Handinnenflächen besitzen wir keine Talgdrüsen.
 Tarsomer nennt man ein Glied in einem gegliederten Fuß eines Insekts.
Tarsomer nennt man ein Glied in einem gegliederten Fuß eines Insekts.
 Tarsus heißt in der Medizin die Fußwurzel, die zusammen mit Mittelfuß und Zehen den Fuß bildet. Insekten- und Spinnenforscher bezeichnen den nach Femur (Schenkel) und Tibia (Schiene) letzten Teil des Beines als Tarsus oder Fuß.
Tarsus heißt in der Medizin die Fußwurzel, die zusammen mit Mittelfuß und Zehen den Fuß bildet. Insekten- und Spinnenforscher bezeichnen den nach Femur (Schenkel) und Tibia (Schiene) letzten Teil des Beines als Tarsus oder Fuß.
 Taxonom nennt man in der Biologie einen Spezialisten für die Taxonomie.
Taxonom nennt man in der Biologie einen Spezialisten für die Taxonomie.
| Taxonomie (von altgriechisch taxis = Ordnung und nomos = Gesetz) heißt allgemein eine Methode, Objekte nach bestimmten Kriterien in Kategorien (Taxa) einzuordnen. Naturwissenschaftler verwenden den Begriff der Taxonomie für eine in der Regel ordnende Klassifikation (z.B.: Klassen, Unterklassen usw.). In der Biologie ist Taxonomie der Name der Teildisziplin, die aufgrund ihrer Eigenschaften versucht, Lebewesen nach dem Grad ihrer Verwandschaft in Domänen, Reiche, Stämme, Klassen, Ordnungen, Familien, Gattungen, Arten (Spezies), Unterarten oder Rassen einzuteilen. |
|
 Technologie im engeren Sinne heißt die Wissenschaft, die Methoden, Werzkeuge und Prozesse untersucht, mit denen Dinge hergestellt, erforscht oder durchgeführt werden können. Die meisten Menschen verstehen aber unter Technologie eher die Gegenstände dieser Forschung. Technologie in diesem Sinne heißt die Summe aller Techniken, Methoden, Arbeitsabläufe, Werkstoffe und Werkzeuge, die zusammenwirken müssen, damit ein bestimmtes Produkt produziert oder eine Arbeit durchgeführt werden kann.
Technologie im engeren Sinne heißt die Wissenschaft, die Methoden, Werzkeuge und Prozesse untersucht, mit denen Dinge hergestellt, erforscht oder durchgeführt werden können. Die meisten Menschen verstehen aber unter Technologie eher die Gegenstände dieser Forschung. Technologie in diesem Sinne heißt die Summe aller Techniken, Methoden, Arbeitsabläufe, Werkstoffe und Werkzeuge, die zusammenwirken müssen, damit ein bestimmtes Produkt produziert oder eine Arbeit durchgeführt werden kann.
 Teildisziplin ist ein Begriff, der sich vom Begriff Disziplin ableitet. In der Wissenschaft verstehen wir darunter aber nicht die Disziplin, die von Schülern und Soldaten gefordert wird, sondern Disziplin ist für Naturwissenschaftler nur ein anderes Wort für Fach oder eine Fachwissenschaft oder ein Teilgebiet des Wissens. Und eine Teildisziplin ist dementsprechend ein Fach innerhalb eines Faches oder eine Fachwissenschaft innerhalb einer Fachwissenschaft oder ein Teilgebiet von einem Teilgebiet des Wissens. So ist die Biologie eine Disziplin innerhalb der Naturwissenschaften und die Ökologie ist eine Teildisziplin der Biologie.
Teildisziplin ist ein Begriff, der sich vom Begriff Disziplin ableitet. In der Wissenschaft verstehen wir darunter aber nicht die Disziplin, die von Schülern und Soldaten gefordert wird, sondern Disziplin ist für Naturwissenschaftler nur ein anderes Wort für Fach oder eine Fachwissenschaft oder ein Teilgebiet des Wissens. Und eine Teildisziplin ist dementsprechend ein Fach innerhalb eines Faches oder eine Fachwissenschaft innerhalb einer Fachwissenschaft oder ein Teilgebiet von einem Teilgebiet des Wissens. So ist die Biologie eine Disziplin innerhalb der Naturwissenschaften und die Ökologie ist eine Teildisziplin der Biologie.
 Tektorialmembran (Membrana tectoria oder Deckmembran) heißt die zellfreie, aber von Kollagen-Fibrillen durchzogene gallertige Deckschicht über den Haarzellen des Corti-Organs auf der Basilarmembran der Hörschnecke im Innenohr.
Tektorialmembran (Membrana tectoria oder Deckmembran) heißt die zellfreie, aber von Kollagen-Fibrillen durchzogene gallertige Deckschicht über den Haarzellen des Corti-Organs auf der Basilarmembran der Hörschnecke im Innenohr.
 Telencephalon (Telenzephalon, Endhirn) nennt man den bei verschiedenen Tierarten sehr unterschiedlich aussehenden vordersten Abschnitt des Gehirns der Wirbeltiere, der sich im Embryo durch eine paarige Ausstülpung zu beiden Seiten hin aus dem vorderen Abschnitt des Prosencephalons bildet. Diese paarig ausgestülpten Teile des Telencephalons bilden die durch den ebenfalls zum Telencephalon gehörenden Balken verbundenen Hemisphären, die in ihrem Innern noch die Seitenventrikel enthalten. Aus jeder Hemisphäre stülpt sich seinerseits rostral der Bulbus olfactorius aus.
Telencephalon (Telenzephalon, Endhirn) nennt man den bei verschiedenen Tierarten sehr unterschiedlich aussehenden vordersten Abschnitt des Gehirns der Wirbeltiere, der sich im Embryo durch eine paarige Ausstülpung zu beiden Seiten hin aus dem vorderen Abschnitt des Prosencephalons bildet. Diese paarig ausgestülpten Teile des Telencephalons bilden die durch den ebenfalls zum Telencephalon gehörenden Balken verbundenen Hemisphären, die in ihrem Innern noch die Seitenventrikel enthalten. Aus jeder Hemisphäre stülpt sich seinerseits rostral der Bulbus olfactorius aus.
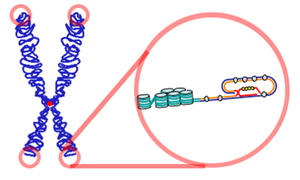
| Telomer heißt das aus endlosen Wiederholungen der immer gleichen kurzen Nukleotidsequenz bestehende Ende eines linearen (nicht kreisförmig geschlossenen) Chromosoms. Telomere schützen die Enden der Chromosomen, denn bei jeder Zellteilung geht von jedem Ende jedes Chromosoms ein Stück verloren. |
 |
| anonym, CC BY-SA 3.0 |
 Telophase nennen Zellbiologen die Phase im Zellzyklus, in der sich der Spindelapparat auflöst, sich an den beiden Zellpolen um die 1-Chromatid-Chromosomen herum neue Kernhüllen bilden und wo in diesen die Chromosomen wieder dekondensieren. Das bedeutet, dass sie wieder ganz lang und dünn werden, sodass man keine einzelnen Chromosomen mehr erkennen kann. Man sieht nur noch eine einzige Masse, die man aufgrund ihrer Färbkarkeit Chromatin nennt.
Telophase nennen Zellbiologen die Phase im Zellzyklus, in der sich der Spindelapparat auflöst, sich an den beiden Zellpolen um die 1-Chromatid-Chromosomen herum neue Kernhüllen bilden und wo in diesen die Chromosomen wieder dekondensieren. Das bedeutet, dass sie wieder ganz lang und dünn werden, sodass man keine einzelnen Chromosomen mehr erkennen kann. Man sieht nur noch eine einzige Masse, die man aufgrund ihrer Färbkarkeit Chromatin nennt.
 Temperatur ist eine innere Eigenschaft eines Gases, einer Flüssigkeit oder eines Körpers, die sich nicht halbiert, wenn man den Gegenstand oder die Menge des Gases bzw. der Flüssigkeit halbiert. Vereinfacht ausgedrückt ist die Temperatur ein Maß für die durchschnittliche kinetische Energie (Geschwindigkeit mal Masse), mit der sich die Atome und Moleküle in Gasen, Flüssigkeiten oder Körpern ungerichtet hin und her bewegen. In Gasen wie der Luft fliegen die Moleküle bei hohen Temperaturen schneller und bei tiefen Temperaturen langsamer. In festen Körpern wie Steinen schwingen die Moleküle bei hohen Temperaturen schneller und bei tiefen Temperaturen langsamer.
Temperatur ist eine innere Eigenschaft eines Gases, einer Flüssigkeit oder eines Körpers, die sich nicht halbiert, wenn man den Gegenstand oder die Menge des Gases bzw. der Flüssigkeit halbiert. Vereinfacht ausgedrückt ist die Temperatur ein Maß für die durchschnittliche kinetische Energie (Geschwindigkeit mal Masse), mit der sich die Atome und Moleküle in Gasen, Flüssigkeiten oder Körpern ungerichtet hin und her bewegen. In Gasen wie der Luft fliegen die Moleküle bei hohen Temperaturen schneller und bei tiefen Temperaturen langsamer. In festen Körpern wie Steinen schwingen die Moleküle bei hohen Temperaturen schneller und bei tiefen Temperaturen langsamer.
 Temperaturoptimum nennt man eine Temperatur oder einen Temperaturbereich, der optimal für einen temperaturabhängigen Prozess wie beispielsweise die Arbeit eines Enzyms ist.
Temperaturoptimum nennt man eine Temperatur oder einen Temperaturbereich, der optimal für einen temperaturabhängigen Prozess wie beispielsweise die Arbeit eines Enzyms ist.
 Temperenz bedeutet Mäßigung. Im Zusammenhang mit Viren ist damit gemeint, dass temperente Viren ihre Baupläne in Genome ihrer Wirtszellen zu integrieren, anstatt sofort den Lytischen Zyklus zu starten.
Temperenz bedeutet Mäßigung. Im Zusammenhang mit Viren ist damit gemeint, dass temperente Viren ihre Baupläne in Genome ihrer Wirtszellen zu integrieren, anstatt sofort den Lytischen Zyklus zu starten.
 temperiert = gemäßigt, nicht zu heiß und nicht zu kalt
temperiert = gemäßigt, nicht zu heiß und nicht zu kalt
 Tempo (von lateinisch tempus = Zeit) bedeutet im normalen Sprachgebrauch einfach Geschwindigkeit oder Schnelligkeit.
Tempo (von lateinisch tempus = Zeit) bedeutet im normalen Sprachgebrauch einfach Geschwindigkeit oder Schnelligkeit.
 Temporallappen nennt man den Schläfenlappen einer Endhirn-Hemisphäre. Hier werden unter anderem die Signale der Hörnerven verarbeitet und interpretiert.
Temporallappen nennt man den Schläfenlappen einer Endhirn-Hemisphäre. Hier werden unter anderem die Signale der Hörnerven verarbeitet und interpretiert.
 temporär = vorübergehend, zeitweilig
temporär = vorübergehend, zeitweilig
 tendenziell = nicht immer, aber überdurchschnittlich häufig zutreffend, nicht generell, aber eher zutreffend, etwas bezweckend, parteilich (z.B.: Berichterstattung)
tendenziell = nicht immer, aber überdurchschnittlich häufig zutreffend, nicht generell, aber eher zutreffend, etwas bezweckend, parteilich (z.B.: Berichterstattung)
 Tenon-Kapsel (Fascia bulbi, Capsula bulbi oder Vagina bulbi) heißt nach dem französischen Chirurgen Jacques-René Tenon benannte Bindegewebe-Kapsel (Faszie) zwischen der äußeren Lederhaut (Sclera) des Auges und dem das Auge in der knöchernen Augenhöhle umgebenden Fettgewebe. Die Tenon-Kapsel umgibt die Sclera und ist mit ihr vorne etwa 2 mm vom Limbus sclerae entfernt und auf der Rückseite im Bereich des Sehnervenaustritts fest verwachsen. Alle Nerven, Muskeln und Blutgefäße treten durch die Tenonsche Kapsel hindurch. Wo die Sehnen der 6 äußeren Augenmuskeln durch die Tenon-Kapsel hindurch ragen, da ragt die Tenon-Kapsel wie oben abgeschnittene Finger eines Handschuhs entlang der Sehnen nach außen. Die Tenon-Kapsel ist wichtig für die Beweglichkeit des Auges, weil sie auf beiden Seiten von leicht verschiebbaren, sehr lockeren Geweben umgeben ist. Zur Sclera hin ist dies das Spatium episcleralis.
Tenon-Kapsel (Fascia bulbi, Capsula bulbi oder Vagina bulbi) heißt nach dem französischen Chirurgen Jacques-René Tenon benannte Bindegewebe-Kapsel (Faszie) zwischen der äußeren Lederhaut (Sclera) des Auges und dem das Auge in der knöchernen Augenhöhle umgebenden Fettgewebe. Die Tenon-Kapsel umgibt die Sclera und ist mit ihr vorne etwa 2 mm vom Limbus sclerae entfernt und auf der Rückseite im Bereich des Sehnervenaustritts fest verwachsen. Alle Nerven, Muskeln und Blutgefäße treten durch die Tenonsche Kapsel hindurch. Wo die Sehnen der 6 äußeren Augenmuskeln durch die Tenon-Kapsel hindurch ragen, da ragt die Tenon-Kapsel wie oben abgeschnittene Finger eines Handschuhs entlang der Sehnen nach außen. Die Tenon-Kapsel ist wichtig für die Beweglichkeit des Auges, weil sie auf beiden Seiten von leicht verschiebbaren, sehr lockeren Geweben umgeben ist. Zur Sclera hin ist dies das Spatium episcleralis.
 Tensor choroideae = Brücke-Muskel
Tensor choroideae = Brücke-Muskel
 Tentorium cerebelli = auch Kleinhirnzelt oder Falx cerebelli genannte, von der Dura mater gebildete, zwischen Großhirn und Kleinhirn in die Schädelhöhle hineinragende Hirnhautfalte, in welcher ein Teil des dorsalen Sinus petrosus (venöser Blutleiter) verläuft (Dr. Ingrid Schütt-Abraham)
Tentorium cerebelli = auch Kleinhirnzelt oder Falx cerebelli genannte, von der Dura mater gebildete, zwischen Großhirn und Kleinhirn in die Schädelhöhle hineinragende Hirnhautfalte, in welcher ein Teil des dorsalen Sinus petrosus (venöser Blutleiter) verläuft (Dr. Ingrid Schütt-Abraham)
 Termination als sogenannter Fachbegriff der Genetik und Molekularbiologie ist in Wirklichkeit gar kein richtiger Fachbegriff, sondern das in vielen biologischen Zusammenhängen einfach aus der englischen Sprache übernommene Wort Termination für Abbruch, Abschluss oder Beendigung. Benutzt wird dieses Pseudofachwort beispielsweise für die Beendigung der Replikation, Transkription und Translation.
Termination als sogenannter Fachbegriff der Genetik und Molekularbiologie ist in Wirklichkeit gar kein richtiger Fachbegriff, sondern das in vielen biologischen Zusammenhängen einfach aus der englischen Sprache übernommene Wort Termination für Abbruch, Abschluss oder Beendigung. Benutzt wird dieses Pseudofachwort beispielsweise für die Beendigung der Replikation, Transkription und Translation.
 Terminator oder genauer Transkriptionsterminator wird in der Genetik eine besondere Nukleotidsequenz in der DNA genannt, die am Ende eines Gens oder bei Bakterien teilweise am Ende eines Operons liegt und zum Abbruch der Transkription führt.
Terminator oder genauer Transkriptionsterminator wird in der Genetik eine besondere Nukleotidsequenz in der DNA genannt, die am Ende eines Gens oder bei Bakterien teilweise am Ende eines Operons liegt und zum Abbruch der Transkription führt.
 Terminologie heißt die Summe aller Termini (Fachausdrücke) eines Fachgebiets. Die in der Biologe übliche Terminologie erklärt dieses Glossar. Gäbe es in der Biologe eine verbindlich festgelegte Terminologie, dann könnte man sie Nomenklatur nennen.
Terminologie heißt die Summe aller Termini (Fachausdrücke) eines Fachgebiets. Die in der Biologe übliche Terminologie erklärt dieses Glossar. Gäbe es in der Biologe eine verbindlich festgelegte Terminologie, dann könnte man sie Nomenklatur nennen.
 Termiten sind staatenbildende, Ameisen-ähnliche Insekten, aber eher mit den Schaben verwandt. Die meisten Termiten sind weniger als 2 Zentimeter lang und sehr hell gefärbt. Fliegen können nur die jungen Königinnen und Könige während des Hochzeitsfluges. Anders als bei Ameisen gibt es in Termiten-Staaten weibliche und männliche Arbeiter und Soldaten und die Königin paart sich immer wieder mit ihrem König. Termiten fressen bevorzugt Gras und Holz. Gerne darf es auch mal ein Holzhaus sein. Ihre Nester bauen sie teilweise in Bäumen, in der Erde oder als spektakuläre, vollklimatisierte Termitenhügel.
Termiten sind staatenbildende, Ameisen-ähnliche Insekten, aber eher mit den Schaben verwandt. Die meisten Termiten sind weniger als 2 Zentimeter lang und sehr hell gefärbt. Fliegen können nur die jungen Königinnen und Könige während des Hochzeitsfluges. Anders als bei Ameisen gibt es in Termiten-Staaten weibliche und männliche Arbeiter und Soldaten und die Königin paart sich immer wieder mit ihrem König. Termiten fressen bevorzugt Gras und Holz. Gerne darf es auch mal ein Holzhaus sein. Ihre Nester bauen sie teilweise in Bäumen, in der Erde oder als spektakuläre, vollklimatisierte Termitenhügel.
 Terra Preta entsteht aus kleinen Holzkohle-Stückchen gemischt mit Fäkalien und Kompost. Hinzu kommen Mikroorganismen, welche das Organische Material zu Pflanzennährstoffen zersetzen, die dann von der riesigen Oberfläche der extrem porösen Holzkohle gebunden werden. Diese schwarze Erde bindet Kohlenstoff für Jahrtausende im Boden und verbessert enorm die Bodenfruchtbarkeit, indem sie Wasser und Nährstoffe bindet. Oft enthält Terra Preta außerdem Keramik-Scherben, die den Mikroorganismen und Pflanzen lebenswichtige Mineralstoffe liefern.
Terra Preta entsteht aus kleinen Holzkohle-Stückchen gemischt mit Fäkalien und Kompost. Hinzu kommen Mikroorganismen, welche das Organische Material zu Pflanzennährstoffen zersetzen, die dann von der riesigen Oberfläche der extrem porösen Holzkohle gebunden werden. Diese schwarze Erde bindet Kohlenstoff für Jahrtausende im Boden und verbessert enorm die Bodenfruchtbarkeit, indem sie Wasser und Nährstoffe bindet. Oft enthält Terra Preta außerdem Keramik-Scherben, die den Mikroorganismen und Pflanzen lebenswichtige Mineralstoffe liefern.
 Terrarium heißt ein relativ kleiner, rundherum geschlossener, aber belüfteter und durch mindestens eine Glasscheibe einsehbarer künstlicher Lebensraum für Pflanzen und wenigstens zeitweise an Land lebende Tiere.
Terrarium heißt ein relativ kleiner, rundherum geschlossener, aber belüfteter und durch mindestens eine Glasscheibe einsehbarer künstlicher Lebensraum für Pflanzen und wenigstens zeitweise an Land lebende Tiere.
 Tertiär ist ein veralteter Name für ein Erdzeitalter, der eigentlich nicht mehr verwendet werden sollte. Das wird er aber und besonders in Schulbüchern. Deshalb sollte man wissen, dass damit die Zeitspanne von 66-2,588 Millionen Jahren vor heute gemeint ist, die man heute in die Systeme Paläogen und Neogen oder in die Serien Paläozän, Eozän, Oligozän, Miozän und Pliozän unterteilt.
Tertiär ist ein veralteter Name für ein Erdzeitalter, der eigentlich nicht mehr verwendet werden sollte. Das wird er aber und besonders in Schulbüchern. Deshalb sollte man wissen, dass damit die Zeitspanne von 66-2,588 Millionen Jahren vor heute gemeint ist, die man heute in die Systeme Paläogen und Neogen oder in die Serien Paläozän, Eozän, Oligozän, Miozän und Pliozän unterteilt.
 Tertiärkonsumenten fressen Sekundärkonsumenten.
Tertiärkonsumenten fressen Sekundärkonsumenten.
 Tertiärstruktur nennt man in der Biochemie die aus allen Sekundärstrukturen bestehende dreidimensionale Faltungsstruktur eines Makromoleküls (meistens eines Proteins).
Tertiärstruktur nennt man in der Biochemie die aus allen Sekundärstrukturen bestehende dreidimensionale Faltungsstruktur eines Makromoleküls (meistens eines Proteins).
 Testosterone sind die wichtigsten männliche Sexualhormone. Sie werden unter dem Einfluss des luteinisierenden Hormons (LH) hauptsächlich in den Hoden produziert. In geringen Mengen wird es auch in den Nebennierenrinden und Eierstöcken produziert. Männer benötigen Testosteron ist nicht nur für die Pubertät, sondern auch für Gesundheit und Ausgeglichenheit. Seine Produktion lässt mit zunehmendem Alter immer stärker nach, kann aber durch Sport gesteigert werden.
Testosterone sind die wichtigsten männliche Sexualhormone. Sie werden unter dem Einfluss des luteinisierenden Hormons (LH) hauptsächlich in den Hoden produziert. In geringen Mengen wird es auch in den Nebennierenrinden und Eierstöcken produziert. Männer benötigen Testosteron ist nicht nur für die Pubertät, sondern auch für Gesundheit und Ausgeglichenheit. Seine Produktion lässt mit zunehmendem Alter immer stärker nach, kann aber durch Sport gesteigert werden.
 Tetanus oder Wundstarrkrampf ist eine oft tödliche Infektionskrankheit. Sie wird vom Bakterium Clostridium tetani verursacht, dessen äußerst widerstandfähige Sporen praktisch überall vorhanden sind. Wenn sie mit dem Schmutz in eine offene Wunde gelangen, werden die Sporen in sauerstoffarmen Umgebungen wieder zu lebenden Bakterien, die Gifte produzieren. Das proteolytische Toxin Tetanospasmin schädigt die muskelsteuernden motorische Nerven und bewirken dadurch schmerzhafte und schließlich oft tödliche Muskelkrämpfe. Gleichzeitig schädigt das Toxin Tetanolysin das Herz.
Tetanus oder Wundstarrkrampf ist eine oft tödliche Infektionskrankheit. Sie wird vom Bakterium Clostridium tetani verursacht, dessen äußerst widerstandfähige Sporen praktisch überall vorhanden sind. Wenn sie mit dem Schmutz in eine offene Wunde gelangen, werden die Sporen in sauerstoffarmen Umgebungen wieder zu lebenden Bakterien, die Gifte produzieren. Das proteolytische Toxin Tetanospasmin schädigt die muskelsteuernden motorische Nerven und bewirken dadurch schmerzhafte und schließlich oft tödliche Muskelkrämpfe. Gleichzeitig schädigt das Toxin Tetanolysin das Herz.
| Tetraeder (genauer: regelmäßiges Tetraeder) heißt ein perfekter (platonischer) Körper, dessen Oberfläche aus vier gleichseitigen Dreiecken als Seitenflächen besteht und dadurch eine dreiseitige Pyramide (Dreieckpyramide) bildet. |

|
| Peter Steinberg, CC BY-SA 3.0 |
Der Thalamus ist als größtes und zentrales Kerngebiet des Zwischenhirns (Diencephalon) die ziemlich zentral unter dem Mittelpunkt des Hirnes auf der Spitze des Hirnstammes (Truncus encephali) gelegene, besonders voluminöse graue Masse beiderseits des Hohlraumes zwischen den Großhirn -Hemisphären. Man unterscheidet in ihm viele separate Kerne mit verschiedenen Funktionen und Verbindungen zu Rückenmark, Kleinhirn (Cerebellum), extrapyramidalem System und fast allen Regionen der Großhirnrinde. Er wird oft "Tor zum Bewußtsein" genannt, weil er eine subkortikale Sammel- und Verknüpfungsstelle für die Sinnessysteme (außer Geruchssinn) und die inneren Sensoren des Körpers mit Umschaltung zur Großhirnrinde ist. Teilweise erzeugt der Thalamus aber auch selbständig Reaktionen auf Empfindungen. Der Thalamus kontrolliert und bewertet alle Erregungen, die von den Sinnesorganen in die Großhirnrinde gehen und fördert oder unterdrückt sie. Das wirkt sich auf die Motorik, die Sinneswahrnehmung und zusammen mit dem limbischen System auf die Empfindungen und damit auf die Gefühle und das Verhalten aus.
 Thallus nennt man die Zellfäden (Vorfäden), pilzähnliche Geflechte aus Zellfäden oder flache Zellhaufen, die bei vielen Moosen aus den Sporen heraus wachsen. Sie dienen der ungeschlechtlichen Fortpflanzung, denn unabhängig von der Form spricht man von einem Vorkeim, aus dem heraus jeweils mehrere neue auch mit bloßem Auge sichtbare Moos-Pflänzchen wachsen, die wie eineiige Zwillinge natürliche Klone sind, weil sie alle den selben haploiden Bauplan besitzen.
Thallus nennt man die Zellfäden (Vorfäden), pilzähnliche Geflechte aus Zellfäden oder flache Zellhaufen, die bei vielen Moosen aus den Sporen heraus wachsen. Sie dienen der ungeschlechtlichen Fortpflanzung, denn unabhängig von der Form spricht man von einem Vorkeim, aus dem heraus jeweils mehrere neue auch mit bloßem Auge sichtbare Moos-Pflänzchen wachsen, die wie eineiige Zwillinge natürliche Klone sind, weil sie alle den selben haploiden Bauplan besitzen.
THC  |
|---|
 |
| anonym, Creative Commons Attribution 3.0 Unported. |
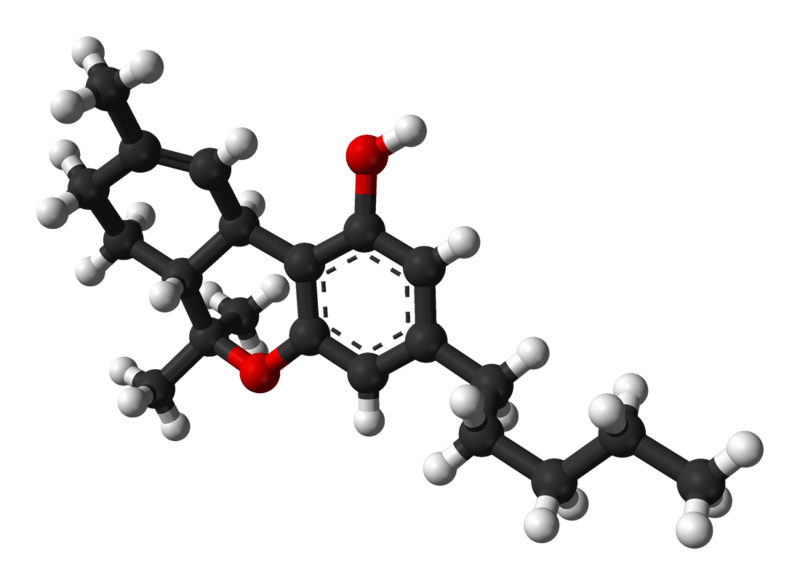
| THC kann unter vielen anderen die Abkürzung sein für Tetrahydrocannabinol oder genauer (-)-Δ-9-trans-Tetrahydrocannabinol. Das ist eine psychoaktive Substanz aus der Gruppe der Cannabinoiden. Auf dieses THC ist die berauschende Wirkung der Hanf-Produkte Marihuana und Haschisch hauptsächlich zurückzuführen. Besonders reich an THC sind die unbefruchteten weiblichen Blütenstände, relativ viel THC enthalten aber auch die Blätter nahe der weiblichen Blüte. Männliche Hanf-Pflanzen haben im Unterschied zu weiblichen einen sehr geringen THC-Gehalt. In den Samen der Hanf-Pflanze ist gar kein THC enthalten. |
 Theoretische Biologie ist der Zweig (die Teildisziplin) der Biologie, der die experimentell oder durch Beobachtung gewonnenen Daten der experimentellen Biologie sammelt, ordnet, zusammenfasst und überprüft und dann auf dieser Basis versucht, gesichertes und neues Wissen zu gewinnen und mit neuen Hypothesen oder Theorien sinnvoll weiterführende Experimente anzuregen. Im Gegensatz zur experimentellen Biologie macht man also in der theoretischen Biologie keine Experimente, aber man braucht eine solide Ausbildung und Erfahrung als experimenteller Biologe, um die Veröffentlichungen der experimentell arbeitenden Biologen zu verstehen und zu erkennen, wo diese schummeln, etwas verschweigen, schlampig gearbeitet haben oder ihre eigenen Daten falsch interpretieren. Anders als die sehr viel ältere und bekanntere Theoretische Physik ist daher die Theoretische Biologie überhaupts nichts für Anfänger. Andererseits ist die ganze Schulbiologie im Wesentlichen nur pseudoexperimentell und daher eigentlich eine Art theoretische Biologie für absolute Anfänger. Daher kann es meines Erachtens nicht schaden, wenn die Lernenden Grundtechniken der Theoretischen Biologie erlernen. Es ist ja generell und nicht nur für die Biologie nützlich, wenn man recherchieren, analysieren, präzise und unmissverständlich formulieren, Wissen grafisch darstellen, korrekt zitieren und von Anderen produzierte Darstellungen legal nutzen kann.
Theoretische Biologie ist der Zweig (die Teildisziplin) der Biologie, der die experimentell oder durch Beobachtung gewonnenen Daten der experimentellen Biologie sammelt, ordnet, zusammenfasst und überprüft und dann auf dieser Basis versucht, gesichertes und neues Wissen zu gewinnen und mit neuen Hypothesen oder Theorien sinnvoll weiterführende Experimente anzuregen. Im Gegensatz zur experimentellen Biologie macht man also in der theoretischen Biologie keine Experimente, aber man braucht eine solide Ausbildung und Erfahrung als experimenteller Biologe, um die Veröffentlichungen der experimentell arbeitenden Biologen zu verstehen und zu erkennen, wo diese schummeln, etwas verschweigen, schlampig gearbeitet haben oder ihre eigenen Daten falsch interpretieren. Anders als die sehr viel ältere und bekanntere Theoretische Physik ist daher die Theoretische Biologie überhaupts nichts für Anfänger. Andererseits ist die ganze Schulbiologie im Wesentlichen nur pseudoexperimentell und daher eigentlich eine Art theoretische Biologie für absolute Anfänger. Daher kann es meines Erachtens nicht schaden, wenn die Lernenden Grundtechniken der Theoretischen Biologie erlernen. Es ist ja generell und nicht nur für die Biologie nützlich, wenn man recherchieren, analysieren, präzise und unmissverständlich formulieren, Wissen grafisch darstellen, korrekt zitieren und von Anderen produzierte Darstellungen legal nutzen kann.
 Theorie nennt man eine vereinfachte Vorstellung (Modell) eines bestimmten Aspekts unserer Welt. Eine Theorie soll einen winzigen Ausschnitt der an sich in ihrer Komplexität und mit ihren vielen für uns nicht wahrnehmbaren Dimensionen unbegreiflichen Natur für uns Menschen anschaulich machen. Theorien beschreiben also die Welt nicht wie sie tatsächlich ist, sondern wie wir sie möglichst gut verstehen oder wenigstens sinnvoll mit ihr umgehen können. Theorien gehen von bestimmten Annahmen aus, die nicht endgültig bewiesen, aber im Laufe der Zeit immer besser belegt oder aber widerlegt werden können. Theorien ermöglichen es uns, bestimmte Beobachtungen oder Ergebnisse von Experimenten vorherzusagen. Mit nicht zutreffenden Vorhersagen lassen sich Theorien auch widerlegen. Von Hypothesen unterscheiden sich Theorien ohne scharfe Grenze dadurch, dass sie schon einige Male durch Beobachtungen oder Experimente nicht wiederlegt, sondern nur bestätigt werden konnten.
Theorie nennt man eine vereinfachte Vorstellung (Modell) eines bestimmten Aspekts unserer Welt. Eine Theorie soll einen winzigen Ausschnitt der an sich in ihrer Komplexität und mit ihren vielen für uns nicht wahrnehmbaren Dimensionen unbegreiflichen Natur für uns Menschen anschaulich machen. Theorien beschreiben also die Welt nicht wie sie tatsächlich ist, sondern wie wir sie möglichst gut verstehen oder wenigstens sinnvoll mit ihr umgehen können. Theorien gehen von bestimmten Annahmen aus, die nicht endgültig bewiesen, aber im Laufe der Zeit immer besser belegt oder aber widerlegt werden können. Theorien ermöglichen es uns, bestimmte Beobachtungen oder Ergebnisse von Experimenten vorherzusagen. Mit nicht zutreffenden Vorhersagen lassen sich Theorien auch widerlegen. Von Hypothesen unterscheiden sich Theorien ohne scharfe Grenze dadurch, dass sie schon einige Male durch Beobachtungen oder Experimente nicht wiederlegt, sondern nur bestätigt werden konnten.
 Therapie nennt man die Behandlung einer Krankheit.
Therapie nennt man die Behandlung einer Krankheit.
 Therapieren nennt man das Behandeln einer Krankheit.
Therapieren nennt man das Behandeln einer Krankheit.
 therapeutisch = für die Behandlung (Therapie) einer Krankheit, die Behandlung (Therapie) einer Krankheit betreffend, der Heilung dienend
therapeutisch = für die Behandlung (Therapie) einer Krankheit, die Behandlung (Therapie) einer Krankheit betreffend, der Heilung dienend
 thermische Energie = Wärmeenergie
thermische Energie = Wärmeenergie
 Thermodynamik oder Wärmelehre heißt das Wissensgebiet, das uns zu erklären versucht, was Wärme, Arbeit im physikalischen Sinne, innere Energien von Stoffen und Ordnung beziehungsweise Unordnung sind, wie sich verschiedene Energieformen und Aggregatzustände in einander umwandeln lassen und was chemische Reaktionen antreibt. Mit Hilfe von 4 Hauptsätzen der Thermodynamik und zahlreichen durch Erfahrung gewonnenen Gleichungen ermöglicht die Thermodynamik für Systeme aus sehr vielen Teilchen Vorhersagen über mögliche bzw. nicht mögliche Änderungen des Systems hinsichtlich seiner Energie und Ordnung. So lässt sich beispielsweise vorhersagen, welche chemischen Reaktionen oder Phasenübergänge spontan ablaufen können, wieviel Wärmeenergie dabei frei wird und wie sich dabei Druck, Temperatur oder Volumen ändern. Dabei ergeben sich Druck und Temperatur als Mittelwerte aus den kinetischen Energien unzähliger Teilchen mit den Vorteilen leichter Messbarkeit und Aussagekraft hinsichtlich des Zustandes eines Systems. Die Thermodynamik erklärt uns die Welt mit vier Hauptsätzen, die längst mathematisch exakt formuliert wurden. Allgemein verständlicher sind sie aber in ihrer ursprünglichen Form als Erfahrungssätze, die aus Beobachtungen abgeleitet wurden. Dazu führt die Thermodynamik seltsame Begriffe wie Entropie, Exergie und Anergie ein und definiert aus der Umgangssprache gewohnte Begriffe wie Temperatur, Wärme und Arbeit auf ungewohnte Weise. Als Anergie wird der Bestandteil einer Energie bezeichnet, der in einem Prozess keine Arbeit verrichten kann. Entsprechend ist Exergie der Anteil einer Energie, der in andere Energieformen umgewandelt werden und dadurch in einem Prozess Arbeit verrichten kann. Die vier Hauptsätze der Gleichgewichtsthermodynamik sind:
Thermodynamik oder Wärmelehre heißt das Wissensgebiet, das uns zu erklären versucht, was Wärme, Arbeit im physikalischen Sinne, innere Energien von Stoffen und Ordnung beziehungsweise Unordnung sind, wie sich verschiedene Energieformen und Aggregatzustände in einander umwandeln lassen und was chemische Reaktionen antreibt. Mit Hilfe von 4 Hauptsätzen der Thermodynamik und zahlreichen durch Erfahrung gewonnenen Gleichungen ermöglicht die Thermodynamik für Systeme aus sehr vielen Teilchen Vorhersagen über mögliche bzw. nicht mögliche Änderungen des Systems hinsichtlich seiner Energie und Ordnung. So lässt sich beispielsweise vorhersagen, welche chemischen Reaktionen oder Phasenübergänge spontan ablaufen können, wieviel Wärmeenergie dabei frei wird und wie sich dabei Druck, Temperatur oder Volumen ändern. Dabei ergeben sich Druck und Temperatur als Mittelwerte aus den kinetischen Energien unzähliger Teilchen mit den Vorteilen leichter Messbarkeit und Aussagekraft hinsichtlich des Zustandes eines Systems. Die Thermodynamik erklärt uns die Welt mit vier Hauptsätzen, die längst mathematisch exakt formuliert wurden. Allgemein verständlicher sind sie aber in ihrer ursprünglichen Form als Erfahrungssätze, die aus Beobachtungen abgeleitet wurden. Dazu führt die Thermodynamik seltsame Begriffe wie Entropie, Exergie und Anergie ein und definiert aus der Umgangssprache gewohnte Begriffe wie Temperatur, Wärme und Arbeit auf ungewohnte Weise. Als Anergie wird der Bestandteil einer Energie bezeichnet, der in einem Prozess keine Arbeit verrichten kann. Entsprechend ist Exergie der Anteil einer Energie, der in andere Energieformen umgewandelt werden und dadurch in einem Prozess Arbeit verrichten kann. Die vier Hauptsätze der Gleichgewichtsthermodynamik sind:
 thermodynamisch = die Thermodynamik betreffend, im Sinne der Thermodynamik
thermodynamisch = die Thermodynamik betreffend, im Sinne der Thermodynamik
 Thermodynamisches System nennt man ein offenes oder geschlossenes System, welches sich durch die Gesetze der Thermodynamik beschreiben lässt. Allerdings muss es sich dabei nicht um ein System in dem Sinne handeln, dass es aus zusammen wirkenden Teilen besteht, die durch ihr Zusammenwirken etwas neues, größeres schaffen. Ein thermodynamisches System kann einfach ein auf Wasser schwimmendes Stück Eis sein oder das Wasser in einem Becher.
Thermodynamisches System nennt man ein offenes oder geschlossenes System, welches sich durch die Gesetze der Thermodynamik beschreiben lässt. Allerdings muss es sich dabei nicht um ein System in dem Sinne handeln, dass es aus zusammen wirkenden Teilen besteht, die durch ihr Zusammenwirken etwas neues, größeres schaffen. Ein thermodynamisches System kann einfach ein auf Wasser schwimmendes Stück Eis sein oder das Wasser in einem Becher.
Physiker und Biologen nennen thermodynamische Systeme offen, wenn sie mit ihren Umgebungen Stoffe UND Energie austauschen können. Geschlossene Systeme können mit ihren Umgebungen nur Energie, aber keine Materie austauschen. Da es keine hundertprozentigen Isolierungen gibt, existieren abgeschlossene Systeme nur theoretisch, denn sie könnten definitionsgemäß mit ihren Umgebungen weder Materie noch Energie austauschen.
 Thermometer nennt man Geräte zur Messung von Temperaturen.
Thermometer nennt man Geräte zur Messung von Temperaturen.
 Thermorezeptoren können in bestimmten Temperatur-Bereichen auf Temperatur-Änderungen oder auf verschiedene Temperaturen unterschiedlich reagieren. Im menschlichen Körper gibt es Thermorezeptoren unter anderem in der Haut, aber nicht in jedem Hautbereich gleich viele. Außerdem unterscheidet man bei 5 bis 40°C aktive Kalt-Rezeptoren von den wesentlich selteneren, bei fast 30°C bis gut 40°C aktiven Warm-Rezeptoren und den erst ab 43°C reagierenden Heiß-Rezeptoren. Thermorezeptoren sind die peripheren Enden pseudounipolarer Neuronen. In den Zellmembranen dieser Dendrit befinden sie Kanäle aus Proteinen, die bei bestimmten Temperaturänderungen ihre Form etwas ändern, sodass sich der Kanal öffnet und eine Depolarisierung bewirkt, die sich in Richtung Axon ausbreitet. Von diesen Kanal-Proteinen gibt es verschiedene, die bei unterschiedlichen Temperaturen reagieren.
Thermorezeptoren können in bestimmten Temperatur-Bereichen auf Temperatur-Änderungen oder auf verschiedene Temperaturen unterschiedlich reagieren. Im menschlichen Körper gibt es Thermorezeptoren unter anderem in der Haut, aber nicht in jedem Hautbereich gleich viele. Außerdem unterscheidet man bei 5 bis 40°C aktive Kalt-Rezeptoren von den wesentlich selteneren, bei fast 30°C bis gut 40°C aktiven Warm-Rezeptoren und den erst ab 43°C reagierenden Heiß-Rezeptoren. Thermorezeptoren sind die peripheren Enden pseudounipolarer Neuronen. In den Zellmembranen dieser Dendrit befinden sie Kanäle aus Proteinen, die bei bestimmten Temperaturänderungen ihre Form etwas ändern, sodass sich der Kanal öffnet und eine Depolarisierung bewirkt, die sich in Richtung Axon ausbreitet. Von diesen Kanal-Proteinen gibt es verschiedene, die bei unterschiedlichen Temperaturen reagieren.
 These nennt man eine Behauptung, die erst noch bewiesen werden müsste oder Widerspruch geradezu herausfordert.
These nennt man eine Behauptung, die erst noch bewiesen werden müsste oder Widerspruch geradezu herausfordert.
 Thiamin = Vitamin B1
Thiamin = Vitamin B1
 Thorax = Brustkorb oder Oberkörper zwischen Hals und Zwerchfell
Thorax = Brustkorb oder Oberkörper zwischen Hals und Zwerchfell
 Thrombenbildung = Entstehung von Thromben als Folge der Blutgerinnung im lebenden Organismus (Dr. Ingrid Schütt-Abraham)
Thrombenbildung = Entstehung von Thromben als Folge der Blutgerinnung im lebenden Organismus (Dr. Ingrid Schütt-Abraham)
 Thromboplastin = Sammelbezeichnung für verschiedene Zwischenprodukte der Blutgerinnung, zu denen das Gewebsthromboplastin (Faktor III), das Plasmathromboplastin (Komplex aus Faktor VIIIa, IXa, Calciumionen und dem Plättchenfaktor 3) sowie das partielle Thromboplastin (Plättchenfaktor 3 selbst) gehören (Dr. Ingrid Schütt-Abraham)
Thromboplastin = Sammelbezeichnung für verschiedene Zwischenprodukte der Blutgerinnung, zu denen das Gewebsthromboplastin (Faktor III), das Plasmathromboplastin (Komplex aus Faktor VIIIa, IXa, Calciumionen und dem Plättchenfaktor 3) sowie das partielle Thromboplastin (Plättchenfaktor 3 selbst) gehören (Dr. Ingrid Schütt-Abraham)
 Thrombose nennt man eine plötzliche Verstopfung eines Blutgefäßes durch verklumpte Blutkörperchen.
Thrombose nennt man eine plötzliche Verstopfung eines Blutgefäßes durch verklumpte Blutkörperchen.
Thrombozyt  |
|---|
| Blausen.com staff, Creative Commons Attribution 3.0 Unported. |
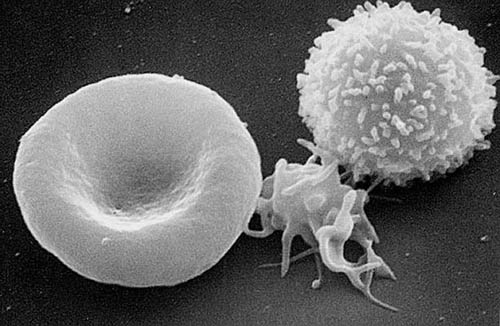
| Thrombozyten oder Blutplättchen heißen die kleinsten Zellen des Blutes. Im Falle einer Blutung setzen sie gerinnungsfördernde Stoffe frei und verbinden sich untereinander sowie mit dem das zu schließende Loch umgebenden Gewebe. Ähnlich wie die Erythrozyten besitzen auch die Blutplättchen keinen Zellkern mehr. Sie entstehen im Knochenmark durch Abschnürung aus Megakaryozyten. Noch nicht aktivierte Thrombozyten haben Durchmesser von 1,5-3,0 µm und sind scheibenartig flach (discoid). Während der Blutgerinnung ändern die Thrombozyten durch Aktivatoren wie ADP, Kollagen, Thromboxan und Thrombin ihre Form (shape change). Es erfolgt eine Vervielfachung der Oberfläche durch Ausstülpung von Pseudopodien. Als normal gelten 150.000-380.000 Thrombozyten pro µl Blut. Die durchschnittliche Lebensdauer von Thrombozyten beträgt acht bis zwölf Tage. Der Abbau erfolgt hauptsächlich in der Milz sowie in der Lunge und, in geringerem Maße, in der Leber. |
 |
| Electron Microscopy Facility at The National Cancer Institute at Frederick, public domain. |
 Thrombus = Blutpfropf (geronnene Blutmasse). Thromben können durch Anlagerung von Blutplättchen (Thrombozyten) an Gefäßwanddefekte entstehen (Abscheidungs- bzw. Agglutinationsthrombus, weißer Thrombus), oder infolge Blutstauung im Gefäß unter Einschluss roter Blutkörperchen (Koagulationsthrombus, roter Thrombus). Häufig kommt es auch zur Mischung von beiden. (Dr. Ingrid Schütt-Abraham)
Thrombus = Blutpfropf (geronnene Blutmasse). Thromben können durch Anlagerung von Blutplättchen (Thrombozyten) an Gefäßwanddefekte entstehen (Abscheidungs- bzw. Agglutinationsthrombus, weißer Thrombus), oder infolge Blutstauung im Gefäß unter Einschluss roter Blutkörperchen (Koagulationsthrombus, roter Thrombus). Häufig kommt es auch zur Mischung von beiden. (Dr. Ingrid Schütt-Abraham)
 Thymin (abgekürzt T) nennt man die Pyrimidin-Base 2,4-Dioxo-5-methylpyrimidin (5-Methyluracil), die mit D-Desoxyribose das Nukleosid Desoxythymidin bildet.
Thymin (abgekürzt T) nennt man die Pyrimidin-Base 2,4-Dioxo-5-methylpyrimidin (5-Methyluracil), die mit D-Desoxyribose das Nukleosid Desoxythymidin bildet.
 Thymidin (abgekürzt T) nennt man verkürzt ein eigentlich Desoxythymidin heißendes, aus der Pyrimidin-Base Thymin und der Pentose (Zucker mit 5 Kohlenstoff-Atomen) D-Desoxyribose gebildetes Nukleosid. Durch Veresterung von Thymidin-Nukleosiden mit Phosphorsäure entstehen die Desoxyribonukleotide Thymidinmonophosphat (dTMP), Thymidindiphosphat (dTDP) und Thymidintriphosphat (dTTP). dTMP ist einer der 4 normalen Bausteine der DNA. RNA enthält statt Thymidin dessen unmethyliertes Derivat Uridin.
Thymidin (abgekürzt T) nennt man verkürzt ein eigentlich Desoxythymidin heißendes, aus der Pyrimidin-Base Thymin und der Pentose (Zucker mit 5 Kohlenstoff-Atomen) D-Desoxyribose gebildetes Nukleosid. Durch Veresterung von Thymidin-Nukleosiden mit Phosphorsäure entstehen die Desoxyribonukleotide Thymidinmonophosphat (dTMP), Thymidindiphosphat (dTDP) und Thymidintriphosphat (dTTP). dTMP ist einer der 4 normalen Bausteine der DNA. RNA enthält statt Thymidin dessen unmethyliertes Derivat Uridin.
 Thymopoese (englisch: B Thymopoiesis) nennt man die im Thymus stattfindende Entwicklung zahlreicher T-Lymphozyten aus wenigen hämopoetischen Vorläuferzellen während einer Wanderung von außen nach innen durch Cortex und Medulla des Thymus. Die früher auch Thymozytopoese genannte Thymopoese ist Teil der T-Lymphopoese. Zu der Entwicklung (Differenzierung) der bei Mäusen common lymphoid progenitors (CLP) und bei Menschen Multi-lymphoid Progenitor (MLP) genannten Vorläuferzellen im Thymus gehört vor allem das Zusammenbasteln eines einzigartigen, also in jedem T-Lymphozyten anderen Gens für einen T-Zell-Rezeptor auf zahlreichen alternativen Fragmenten. Dabei werden schon im Cortex alle Thymozyten eliminiert, deren T-Zell-Rezeptor - ß-Kette unbrauchbar ist. Danach wird ebenfalls noch im Cortex des Thymus getestet, ob ein intakter T-Zell-Rezeptor auch in der Lage ist, mit den MHC-Präsentiertellern auf den Oberflächen anderer Zellen zu interagieren. Thymozyten sind nutzlos und müssen sterben, wenn ihre T-Zell-Rezeptoren das nicht können. Wenn sie es können, dann gibt es zwei Möglichkeiten. Erkennen die T-Zell-Rezeptoren eines Thymozyten zu den auf fast allen Körperzelltypen zu findenden MHC-1-Präsentiertellern, dann wird er eine T-Killerzelle. Passt hingegen der T-Zell-Rezeptor des Thymozyten zu den nur von Antigen präsentierenden Zellen gezeigten MHC-2-Präsentiertellern, dann könnte aus dem Thymozyten eine T-Helferzelle werden. Vorher müssen allerdings alle noch lebenden Thymozyten nach Bestehen der Positiv-Tests einen noch viel wichtigeren Negativ-Test bestehen, der hauptsächlich in der Medulla des Thymus stattfindet. Sie werden nämlich aktiv durch induzierte Apoptose getötet, wenn sie irgendein körpereigenes Antigen erkennen. Ansonsten könnten sie nämlich normle Zellen des eigenen Körpers angreifen und so eine Autoimmunkrankheit verursachen. Deshalb werden in der Medulla des Thymus extra möglichst alle im Körper vorkommenden Peptide produziert und auf Zelloberflächen präsentiert. Nach Bestehen dieser Tests dürfen die ehemaligen Thymozyten als fertige, reife T-Lymphozyten den Thymus verlassen und in den Lymphknoten auf ihre Aktivierung warten.
Thymopoese (englisch: B Thymopoiesis) nennt man die im Thymus stattfindende Entwicklung zahlreicher T-Lymphozyten aus wenigen hämopoetischen Vorläuferzellen während einer Wanderung von außen nach innen durch Cortex und Medulla des Thymus. Die früher auch Thymozytopoese genannte Thymopoese ist Teil der T-Lymphopoese. Zu der Entwicklung (Differenzierung) der bei Mäusen common lymphoid progenitors (CLP) und bei Menschen Multi-lymphoid Progenitor (MLP) genannten Vorläuferzellen im Thymus gehört vor allem das Zusammenbasteln eines einzigartigen, also in jedem T-Lymphozyten anderen Gens für einen T-Zell-Rezeptor auf zahlreichen alternativen Fragmenten. Dabei werden schon im Cortex alle Thymozyten eliminiert, deren T-Zell-Rezeptor - ß-Kette unbrauchbar ist. Danach wird ebenfalls noch im Cortex des Thymus getestet, ob ein intakter T-Zell-Rezeptor auch in der Lage ist, mit den MHC-Präsentiertellern auf den Oberflächen anderer Zellen zu interagieren. Thymozyten sind nutzlos und müssen sterben, wenn ihre T-Zell-Rezeptoren das nicht können. Wenn sie es können, dann gibt es zwei Möglichkeiten. Erkennen die T-Zell-Rezeptoren eines Thymozyten zu den auf fast allen Körperzelltypen zu findenden MHC-1-Präsentiertellern, dann wird er eine T-Killerzelle. Passt hingegen der T-Zell-Rezeptor des Thymozyten zu den nur von Antigen präsentierenden Zellen gezeigten MHC-2-Präsentiertellern, dann könnte aus dem Thymozyten eine T-Helferzelle werden. Vorher müssen allerdings alle noch lebenden Thymozyten nach Bestehen der Positiv-Tests einen noch viel wichtigeren Negativ-Test bestehen, der hauptsächlich in der Medulla des Thymus stattfindet. Sie werden nämlich aktiv durch induzierte Apoptose getötet, wenn sie irgendein körpereigenes Antigen erkennen. Ansonsten könnten sie nämlich normle Zellen des eigenen Körpers angreifen und so eine Autoimmunkrankheit verursachen. Deshalb werden in der Medulla des Thymus extra möglichst alle im Körper vorkommenden Peptide produziert und auf Zelloberflächen präsentiert. Nach Bestehen dieser Tests dürfen die ehemaligen Thymozyten als fertige, reife T-Lymphozyten den Thymus verlassen und in den Lymphknoten auf ihre Aktivierung warten.
 Thymozyt nennt man eine die Thymopoese durchlaufende hämopoetische Vorläuferzelle im Thymus.
Thymozyt nennt man eine die Thymopoese durchlaufende hämopoetische Vorläuferzelle im Thymus.
 Thymus oder Thymusdrüse heißt ein hinter dem Brustbein liegendes Organ des Lymphsystems, in dem die T-Lymphopoese abgeschlossen wird. Danach werden im Thymus möglichst alle T-Lymphozyten getötet, die keine MHC-Präsentierteller erkennen oder die sich gegen Antigene des eigenen Körpers richten würden. Außerdem beeinflusst der Thymus hormonell das neuro-endokrine System, das Körperwachstum und den Knochen-Stoffwechsel. Er wächst bis zum Eintritt der Geschlechtsreife und bildet sich dann zurück bzw. wandelt sich in einen Fettkörper um. (Thymus = bei Kalb und Lamm auch Bries genanntes, im ventralen Halsbereich gelegenes drüsenähnliches lymphatisches Organ, das für den Aufbau der Immunabwehr bei Jungtieren erforderlich ist. Der bei der Geburt von Rindern entlang der Speiseröhre verlaufende, stark ausgeprägte Thymus bildet sich mit zunehmendem Alter zurück und ist bei adulten Tieren in der Regel nicht mehr vorhanden - Dr. Ingrid Schütt-Abraham)
Thymus oder Thymusdrüse heißt ein hinter dem Brustbein liegendes Organ des Lymphsystems, in dem die T-Lymphopoese abgeschlossen wird. Danach werden im Thymus möglichst alle T-Lymphozyten getötet, die keine MHC-Präsentierteller erkennen oder die sich gegen Antigene des eigenen Körpers richten würden. Außerdem beeinflusst der Thymus hormonell das neuro-endokrine System, das Körperwachstum und den Knochen-Stoffwechsel. Er wächst bis zum Eintritt der Geschlechtsreife und bildet sich dann zurück bzw. wandelt sich in einen Fettkörper um. (Thymus = bei Kalb und Lamm auch Bries genanntes, im ventralen Halsbereich gelegenes drüsenähnliches lymphatisches Organ, das für den Aufbau der Immunabwehr bei Jungtieren erforderlich ist. Der bei der Geburt von Rindern entlang der Speiseröhre verlaufende, stark ausgeprägte Thymus bildet sich mit zunehmendem Alter zurück und ist bei adulten Tieren in der Regel nicht mehr vorhanden - Dr. Ingrid Schütt-Abraham)
 TRH = Thyreotropin-Releasinghormon
TRH = Thyreotropin-Releasinghormon
 TSH = Thyroidea stimulierendes Hormon
TSH = Thyroidea stimulierendes Hormon
 Tibia nennt man bei Säugetieren das Schienbein und bei Gliederfüßern den auch Schiene genannten vierten Teil des Beines zwischen Femur und Tarsus.
Tibia nennt man bei Säugetieren das Schienbein und bei Gliederfüßern den auch Schiene genannten vierten Teil des Beines zwischen Femur und Tarsus.
 Tiefwurzler dringen mit einer pfahlartigen Hauptwurzel in tiefere Bodenschichten vor. Von der Hauptwurzel dieser Pflanzen zweigen zahlreiche dünnere Seitenwurzeln ab.
Tiefwurzler dringen mit einer pfahlartigen Hauptwurzel in tiefere Bodenschichten vor. Von der Hauptwurzel dieser Pflanzen zweigen zahlreiche dünnere Seitenwurzeln ab.
 Tiere sind bewegungsfähige Lebewesen, die andere Lebewesen fressen müssen, weil sie anders als Pflanzen und bestimmte Bakterien keine anderen Energie-Quellen wie das Sonnenlicht nutzen können. Ob auch Einzeller mit dieser Eigenschaft zu den Tieren gezählt werden können, ist, wie in der Biologie üblich, umstritten. Ansonsten bestehen Tiere aus vielen Zellen bestehen und bilden im Gegensatz zu Pilzen mehrschichtige Gewebe.
Tiere sind bewegungsfähige Lebewesen, die andere Lebewesen fressen müssen, weil sie anders als Pflanzen und bestimmte Bakterien keine anderen Energie-Quellen wie das Sonnenlicht nutzen können. Ob auch Einzeller mit dieser Eigenschaft zu den Tieren gezählt werden können, ist, wie in der Biologie üblich, umstritten. Ansonsten bestehen Tiere aus vielen Zellen bestehen und bilden im Gegensatz zu Pilzen mehrschichtige Gewebe.
 Tierart nennt man eine Spezies von Tieren.
Tierart nennt man eine Spezies von Tieren.
 tierisch = Tiere betreffend, zu Tieren gehörend, aus Tieren stammend, bestehend oder gewonnen, aufgebaut wie eine Zelle eines Tieres
tierisch = Tiere betreffend, zu Tieren gehörend, aus Tieren stammend, bestehend oder gewonnen, aufgebaut wie eine Zelle eines Tieres
 Tierkörperbeseitigungsanstalt = traditioneller Name für die Einrichtungen, in denen verendete oder notgetötete Tiere, bei der Fleischuntersuchung zum menschlichen Genuss untauglich beurteilte Tiere und Tierkörperteile oder sonstige gefährliche tierische Abfallprodukte beseitigt und in ihnen enthaltene Krankheitserreger wie Parasiten, Bakterien oder Viren durch Erhitzung unter Druck unschädlich gemacht werden (Dr. Ingrid Schütt-Abraham)
Tierkörperbeseitigungsanstalt = traditioneller Name für die Einrichtungen, in denen verendete oder notgetötete Tiere, bei der Fleischuntersuchung zum menschlichen Genuss untauglich beurteilte Tiere und Tierkörperteile oder sonstige gefährliche tierische Abfallprodukte beseitigt und in ihnen enthaltene Krankheitserreger wie Parasiten, Bakterien oder Viren durch Erhitzung unter Druck unschädlich gemacht werden (Dr. Ingrid Schütt-Abraham)
 Tierseuchenkasse = Von den Ländern eingerichtete Körperschaften oder Anstalten des Öffentlichen Rechts, in der die landwirtschaftlichen Tierhaltungsbetriebe gegen anzeigepflichtige Tierseuchen zwangsversichert sind. Der von den Landwirten zu zahlende Beitrag hängt von der Zahl und Art der gehaltenen Tiere ab; im Falle der amtlichen Feststellung einer anzeigepflichtigen Tierseuche werden die Landwirte von den Tierseuchenkassen entschädigt (Dr. Ingrid Schütt-Abraham)
Tierseuchenkasse = Von den Ländern eingerichtete Körperschaften oder Anstalten des Öffentlichen Rechts, in der die landwirtschaftlichen Tierhaltungsbetriebe gegen anzeigepflichtige Tierseuchen zwangsversichert sind. Der von den Landwirten zu zahlende Beitrag hängt von der Zahl und Art der gehaltenen Tiere ab; im Falle der amtlichen Feststellung einer anzeigepflichtigen Tierseuche werden die Landwirte von den Tierseuchenkassen entschädigt (Dr. Ingrid Schütt-Abraham)
| Tight Junction, Zonula occludens oder Schlussleiste heißt eine aus schmalen Bändern von Membranproteinen bestehende dichte Verbindung zwischen einer Epithelzelle und ihren Zellnachbarn. Indem sie den Zellzwischenraum verschließt, behindert sie den Verlust eigener Moleküle sowie das Eindringen anderer Lebewesen in einen vielzelligen Organismus. Außerdem fixieren tight junctions Zellen in ihren Positionen und Orientierungen. |
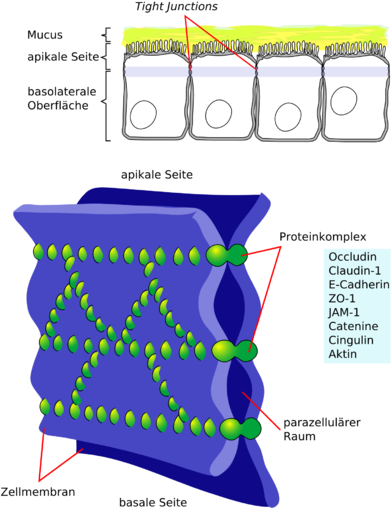
|
| Mariana Ruiz, Public domain |
 Tight Junction, Zonula occludens oder Schlussleiste heißt eine aus schmalen Bändern von Membranproteinen bestehende dichte Verbindung zwischen einer Epithelzelle und ihren Zellnachbarn. In sie den Zellzwischenraum verschließt, behindert sie den Verlust eigener Moleküle sowie das Eindringen anderer Lebewesen in einen vielzelligen Organismus. Außerdem fixieren tight junctions Zellen in ihren Positionen und Orientierungen.
Tight Junction, Zonula occludens oder Schlussleiste heißt eine aus schmalen Bändern von Membranproteinen bestehende dichte Verbindung zwischen einer Epithelzelle und ihren Zellnachbarn. In sie den Zellzwischenraum verschließt, behindert sie den Verlust eigener Moleküle sowie das Eindringen anderer Lebewesen in einen vielzelligen Organismus. Außerdem fixieren tight junctions Zellen in ihren Positionen und Orientierungen.
 Titer = Konzentration einer Substanz, eines Antikörpers oder eines infektiösen Agens.
Titer = Konzentration einer Substanz, eines Antikörpers oder eines infektiösen Agens.
 Titration = Konzentrationsbestimmung
Titration = Konzentrationsbestimmung
 TME = Abkürzung für "Transmissible Mink Encephalopathy", die übertragbare schwammförmige Enzephalopathie der Nerze, eine zu den TSE-Erkrankungen bzw. Prionkrankheiten gehörende Gehirnerkrankung der Nerze (Dr. Ingrid Schütt-Abraham)
TME = Abkürzung für "Transmissible Mink Encephalopathy", die übertragbare schwammförmige Enzephalopathie der Nerze, eine zu den TSE-Erkrankungen bzw. Prionkrankheiten gehörende Gehirnerkrankung der Nerze (Dr. Ingrid Schütt-Abraham)
 Tochterzellen nennt man die beiden Zellen, die bei einer Zellteilung aus einer Zelle entstehen.
Tochterzellen nennt man die beiden Zellen, die bei einer Zellteilung aus einer Zelle entstehen.
 Tod heißt das Ergebnis bzw. der Endzustand des Sterbeprozesses, wenn sich in einem ehemaligen Organismus keinerlei Lebensprozesse mehr abspielen. Man spricht allerdings im übertragenen Sinne auch vom sozialen Tod einsamer Menschen, vom Tod der Persönlichkeit im Endzustand der Alzheimer-Krankheit oder vom Hirntod, wenn man nicht wirklich toten Menschen Organe entnehmen möchte, um das Leben todkranker Patienten zu retten, deren Gehirne als Sitz ihrer Persönlichkeit noch gut funktionieren, während die Gehirne von Hirntoten vermeintlich irreperabel so stark geschädigt sein sollen, dass von ihren Persönlichkeiten nichts mehr übrig ist.
Tod heißt das Ergebnis bzw. der Endzustand des Sterbeprozesses, wenn sich in einem ehemaligen Organismus keinerlei Lebensprozesse mehr abspielen. Man spricht allerdings im übertragenen Sinne auch vom sozialen Tod einsamer Menschen, vom Tod der Persönlichkeit im Endzustand der Alzheimer-Krankheit oder vom Hirntod, wenn man nicht wirklich toten Menschen Organe entnehmen möchte, um das Leben todkranker Patienten zu retten, deren Gehirne als Sitz ihrer Persönlichkeit noch gut funktionieren, während die Gehirne von Hirntoten vermeintlich irreperabel so stark geschädigt sein sollen, dass von ihren Persönlichkeiten nichts mehr übrig ist.
 Toleranz bedeutet zulässige oder akzeptable Abweichung bzw. das Akzeptieren von etwas Abweichendem oder andersartigem bzw. Gewöhnung an etwas eigentlich Schädliches.
Toleranz bedeutet zulässige oder akzeptable Abweichung bzw. das Akzeptieren von etwas Abweichendem oder andersartigem bzw. Gewöhnung an etwas eigentlich Schädliches.
 Toleranzbereich hinsichtlich eines abiotischen Umweltfaktors heißt der Wertebereich, den eine Spezies gut bis gerade noch überleben kann, falls sie nicht zusätzlich der Konkurrenz einer anderen Spezies ausgesetzt ist.
Toleranzbereich hinsichtlich eines abiotischen Umweltfaktors heißt der Wertebereich, den eine Spezies gut bis gerade noch überleben kann, falls sie nicht zusätzlich der Konkurrenz einer anderen Spezies ausgesetzt ist.
 Toleranzkurven oder Optimumskurven zeigen für einen Parameter wie die Wachstumsrate, Überlebensrate, Fortpflanzungsrate oder allgemein Vitalität, wie gut bzw. schlecht es einer Spezies mit den unterschiedlichen Ausprägungen eines bestimmten abiotischen Umweltfaktors geht.
Toleranzkurven oder Optimumskurven zeigen für einen Parameter wie die Wachstumsrate, Überlebensrate, Fortpflanzungsrate oder allgemein Vitalität, wie gut bzw. schlecht es einer Spezies mit den unterschiedlichen Ausprägungen eines bestimmten abiotischen Umweltfaktors geht.
 tolerieren = aushalten, dulden, zulassen
tolerieren = aushalten, dulden, zulassen
 tonischer Krampf = Dauerkrampf, Starrkrampf (Dr. Ingrid Schütt-Abraham)
tonischer Krampf = Dauerkrampf, Starrkrampf (Dr. Ingrid Schütt-Abraham)
 Tonoplast nennen Botaniker die Membran, welche eine Vakuole vom Zytoplasma abgrenzt.
Tonoplast nennen Botaniker die Membran, welche eine Vakuole vom Zytoplasma abgrenzt.
 Tonsilla lingualis = Zungenmandel, ein in der Schleimhaut des Zungengrundes liegendes Organ des lymphoretikulären Sytems (Dr. Ingrid Schütt-Abraham)
Tonsilla lingualis = Zungenmandel, ein in der Schleimhaut des Zungengrundes liegendes Organ des lymphoretikulären Sytems (Dr. Ingrid Schütt-Abraham)
 Tonus = Spannungszustand beispielsweise eines Muskels
Tonus = Spannungszustand beispielsweise eines Muskels
 Topoisomerasen sind Enzyme, welche das Ausmaß der Verdrehung oder Verknotung (Topologie) von DNA-Molekülen verändern können. Sie können Verknotungen lösen und Spannungen auf oder abbauen, indem sie Einzel- oder DNA-Doppelstrang durchschneiden und wieder zusammenkleben.
Topoisomerasen sind Enzyme, welche das Ausmaß der Verdrehung oder Verknotung (Topologie) von DNA-Molekülen verändern können. Sie können Verknotungen lösen und Spannungen auf oder abbauen, indem sie Einzel- oder DNA-Doppelstrang durchschneiden und wieder zusammenkleben.
 Torf heißt ein getrocknet gut brennbares und als Substrat im Gemüseanbau kaum verzichtbares organisches Sediment aus zumindest unvollständig zersetzten Resten abgestorbener Pflanzenteile.
Torf heißt ein getrocknet gut brennbares und als Substrat im Gemüseanbau kaum verzichtbares organisches Sediment aus zumindest unvollständig zersetzten Resten abgestorbener Pflanzenteile.
 Torfmoos heißt eine Torf bildende Moos-Spezies.
Torfmoos heißt eine Torf bildende Moos-Spezies.
 Totholz nennt man in der Ökologie alle abgestorbenen Bäume und Äste. Das ist etwas irreführend, weil Holz ähnlich wie unsere Haare und Fingernägel auch in lebendigen Bäumen nicht mehr lebt.
Totholz nennt man in der Ökologie alle abgestorbenen Bäume und Äste. Das ist etwas irreführend, weil Holz ähnlich wie unsere Haare und Fingernägel auch in lebendigen Bäumen nicht mehr lebt.
 Totipotent oder omnipotent heißt allgemein allmächtig. In der Biologie nennt man so Stammzellen, die noch das Potential haben, alle Zelltypen des Körpers zu bilden. Man findet sie in frühen Embryonen, wo aus ihnen noch alle embryonalen und extraembryonalen Gewebe entstehen.
Totipotent oder omnipotent heißt allgemein allmächtig. In der Biologie nennt man so Stammzellen, die noch das Potential haben, alle Zelltypen des Körpers zu bilden. Man findet sie in frühen Embryonen, wo aus ihnen noch alle embryonalen und extraembryonalen Gewebe entstehen.
 Totipotenz oder Omnipotenz nennt man den Zustand, totipotent zu sein.
Totipotenz oder Omnipotenz nennt man den Zustand, totipotent zu sein.
 Toxin nannten schon die antiken Griechen eine von Lebewesen produzierte giftige Substanz.
Toxin nannten schon die antiken Griechen eine von Lebewesen produzierte giftige Substanz.
 Trabekelwerk = Reticulum trabeculare sclerae
Trabekelwerk = Reticulum trabeculare sclerae
 Tractus = Nervenbahn, Leitungsbahn, zu Bündeln zusammengefasste Nervenfasern
Tractus = Nervenbahn, Leitungsbahn, zu Bündeln zusammengefasste Nervenfasern
 Tractus corticospinalis = Pyramidenbahn, motorische Leitungsbahn, die von den motorischen Zentren der Großhirnrinde bis zu den motorischen Ventralhornzellen des Rückenmarks verläuft und deren größter Teil der Fasern im caudalen Abschnitt der Medulla oblongata auf die Gegenseite kreuzt. Die Pyramidenbahn reicht beim Fleischfresser bis ins Kreuzmark, beim Wiederkäuer jedoch nur bis in das Halsmark (Dr. Ingrid Schütt-Abraham)
Tractus corticospinalis = Pyramidenbahn, motorische Leitungsbahn, die von den motorischen Zentren der Großhirnrinde bis zu den motorischen Ventralhornzellen des Rückenmarks verläuft und deren größter Teil der Fasern im caudalen Abschnitt der Medulla oblongata auf die Gegenseite kreuzt. Die Pyramidenbahn reicht beim Fleischfresser bis ins Kreuzmark, beim Wiederkäuer jedoch nur bis in das Halsmark (Dr. Ingrid Schütt-Abraham)
 Der Tractus olfactorius lateralis ist ein auf der Außenseite des Pars basalis rhinencephali kaudal aus dem Bulbus olfactorius heraus ziehendes, dickes Nervenbündel.
Der Tractus olfactorius lateralis ist ein auf der Außenseite des Pars basalis rhinencephali kaudal aus dem Bulbus olfactorius heraus ziehendes, dickes Nervenbündel.
 Der Tractus olfactorius medialis ist ein auf der Unterseite des Pars basalis rhinencephali kaudal aus dem Bulbus olfactorius heraus ziehendes, dickes Nervenbündel.
Der Tractus olfactorius medialis ist ein auf der Unterseite des Pars basalis rhinencephali kaudal aus dem Bulbus olfactorius heraus ziehendes, dickes Nervenbündel.
 Tractus rubrospinalis = auch Monakowsches Bündel genannte, zum extrapyramidalen System gehörende Leitungsbahn, die vom Nucleus ruber im Dach des Mittelhirns ausgeht, unmittelbar nach ihrem Ursprung auf die andere Körperseite kreuzt und dort unterhalb der Pyramidenbahn je nach Tierart bis in das Kreuzmark verläft (Dr. Ingrid Schütt-Abraham)
Tractus rubrospinalis = auch Monakowsches Bündel genannte, zum extrapyramidalen System gehörende Leitungsbahn, die vom Nucleus ruber im Dach des Mittelhirns ausgeht, unmittelbar nach ihrem Ursprung auf die andere Körperseite kreuzt und dort unterhalb der Pyramidenbahn je nach Tierart bis in das Kreuzmark verläft (Dr. Ingrid Schütt-Abraham)
 Tractus vestibulospinalis = Dem Nucleus vestibularis des Hirnstamms entspringende motorische Leitungsbahn, die ungekreuzt im Ventralstrang des Rückenmarks zu den Motoneuronen der Nacken- und Rückenmuskulatur zieht (Dr. Ingrid Schütt-Abraham)
Tractus vestibulospinalis = Dem Nucleus vestibularis des Hirnstamms entspringende motorische Leitungsbahn, die ungekreuzt im Ventralstrang des Rückenmarks zu den Motoneuronen der Nacken- und Rückenmuskulatur zieht (Dr. Ingrid Schütt-Abraham)
 Trachee heißt eine Röhre. Gement ist damit beispielsweise bei Insekten eine Luftröhre und bei Pflanzeen eine Wasserleitung.
Trachee heißt eine Röhre. Gement ist damit beispielsweise bei Insekten eine Luftröhre und bei Pflanzeen eine Wasserleitung.
 Tränendrüse (lateinisch: Glandula lacrimalis) ist eine in der äußeren Hälfte des oberen Augenlids liegende Drüse, die den größten Teil (rund einen halben Liter täglich) der Tränenflüssigkeit produziert. Diese enthält Ionen und viele unterschiedliche Proteine. Ihr Sekret wird über 6-12 Ausführungsgänge in das Gewölbe des Bindehautsacks (Fornix conjunctivae) geleitet durch Lidschläge auf der Hornhaut (Cornea) verteilt. Die Tränenflüssigkeit hält die Cornea feucht und ernährt sie. Der Abwehr von Krankheitserregern dienen Lysozym und Immunglobulin A. Außer den großen gibt es im menschlichen Augenlid noch andere Tränendrüsen. Die Krause- und Wolfring-Drüsen produzieren rund 5% der wässrigen Tränenflüssigkeit. Die Meibom-Drüsen und die Zeis-Drüsen an den Haarbälgen der Wimpern sind spezialisierte Talgdrüsen. Die Moll-Drüsen vor den Wimpern sind spezialisierte Schweißdrüsen, sollen aber ebenfalls ein Sekret mit Lipiden produzieren.
Tränendrüse (lateinisch: Glandula lacrimalis) ist eine in der äußeren Hälfte des oberen Augenlids liegende Drüse, die den größten Teil (rund einen halben Liter täglich) der Tränenflüssigkeit produziert. Diese enthält Ionen und viele unterschiedliche Proteine. Ihr Sekret wird über 6-12 Ausführungsgänge in das Gewölbe des Bindehautsacks (Fornix conjunctivae) geleitet durch Lidschläge auf der Hornhaut (Cornea) verteilt. Die Tränenflüssigkeit hält die Cornea feucht und ernährt sie. Der Abwehr von Krankheitserregern dienen Lysozym und Immunglobulin A. Außer den großen gibt es im menschlichen Augenlid noch andere Tränendrüsen. Die Krause- und Wolfring-Drüsen produzieren rund 5% der wässrigen Tränenflüssigkeit. Die Meibom-Drüsen und die Zeis-Drüsen an den Haarbälgen der Wimpern sind spezialisierte Talgdrüsen. Die Moll-Drüsen vor den Wimpern sind spezialisierte Schweißdrüsen, sollen aber ebenfalls ein Sekret mit Lipiden produzieren.
 Tränenfilm heißt die bei jedem Lidschlag neu aufgebaute Summe dreier Schichten unterschiedlicher Tränenflüssigkeiten auf der Augenhornhaut. Der Tränenfilm ernährt die Augenhornhaut, schützt sie vor Austrocknung und Krankheitserregern und verbessert entscheidend ihre optischen Eigenschaften, weil er fast alle Unebenheiten der Augenhornhaut ausgleicht. Die von den Becherzellen produzierte unterste Schicht des Tränenfilms ist eine Schleimschicht direkt auf der Augenhornhaut. Die mittlere ist die dickste Schicht und besteht aus den wässigrigen Sekreten der großen Tränendrüsen, der Krause-Drüsen sowie der Wolfring-Drüsen. Die vor Austrockung schützende oberste Schicht des Tränenfilms bilden Lipide, die von den akzessorischen (zusätzlichen) Tränendrüsen Meibom-Drüsen, Moll-Drüsen und Zeis-Drüsen produziert werden.
Tränenfilm heißt die bei jedem Lidschlag neu aufgebaute Summe dreier Schichten unterschiedlicher Tränenflüssigkeiten auf der Augenhornhaut. Der Tränenfilm ernährt die Augenhornhaut, schützt sie vor Austrocknung und Krankheitserregern und verbessert entscheidend ihre optischen Eigenschaften, weil er fast alle Unebenheiten der Augenhornhaut ausgleicht. Die von den Becherzellen produzierte unterste Schicht des Tränenfilms ist eine Schleimschicht direkt auf der Augenhornhaut. Die mittlere ist die dickste Schicht und besteht aus den wässigrigen Sekreten der großen Tränendrüsen, der Krause-Drüsen sowie der Wolfring-Drüsen. Die vor Austrockung schützende oberste Schicht des Tränenfilms bilden Lipide, die von den akzessorischen (zusätzlichen) Tränendrüsen Meibom-Drüsen, Moll-Drüsen und Zeis-Drüsen produziert werden.
 Transfer vom lateinischen transferre = hinüberbringen bedeutet Übertragung.
Transfer vom lateinischen transferre = hinüberbringen bedeutet Übertragung.
 Transferasen katalysieren die Übertragung funktioneller Gruppen (z.: Methyl-, Amino- oder Phosphorylgruppen) von einem Substrat auf ein anderes. Beispielsweise katalysiert die Hexokinase (2.7.1.1) im 1. Schritt der Glykolyse die Übertragung einer Phosphatgruppe von ATP auf Glucose. Die Enzymklasse der Transferasen wird noch einmal unterteilt in der Subklassen: Kinasen, Acetyl-, Methyl- und Aminotransferasen sowie Polymerasen.
Transferasen katalysieren die Übertragung funktioneller Gruppen (z.: Methyl-, Amino- oder Phosphorylgruppen) von einem Substrat auf ein anderes. Beispielsweise katalysiert die Hexokinase (2.7.1.1) im 1. Schritt der Glykolyse die Übertragung einer Phosphatgruppe von ATP auf Glucose. Die Enzymklasse der Transferasen wird noch einmal unterteilt in der Subklassen: Kinasen, Acetyl-, Methyl- und Aminotransferasen sowie Polymerasen.
 Transformation bedeutet Umwandlung oder Umformung. In der Molekularbiologie versteht man darunter speziell die Umwandlung von Bakterien oder Eukaryontenzellen durch die Übertragung fremder DNA.
Transformation bedeutet Umwandlung oder Umformung. In der Molekularbiologie versteht man darunter speziell die Umwandlung von Bakterien oder Eukaryontenzellen durch die Übertragung fremder DNA.
 transformieren = eine Transformation durchführen
transformieren = eine Transformation durchführen
 Transgen nennt man einen Organismus, dem ein zusätzliches, für seine Spezies fremdes Gen mit Hilfe der Gentechnik in alle Körperzellen eingebaut wurde.
Transgen nennt man einen Organismus, dem ein zusätzliches, für seine Spezies fremdes Gen mit Hilfe der Gentechnik in alle Körperzellen eingebaut wurde.
 transkribieren = eine Transkription durchführen
transkribieren = eine Transkription durchführen
 Transkript nennt man das Produkt einer Übersetzung von einer Sprache oder Darstellungsform in eine andere. In der Genetik meint man damit eine RNA-Kopie eines Teils der in der DNA kodierten Erbinformation.
Transkript nennt man das Produkt einer Übersetzung von einer Sprache oder Darstellungsform in eine andere. In der Genetik meint man damit eine RNA-Kopie eines Teils der in der DNA kodierten Erbinformation.
|
Transkription heißt der Vorgang der Produktion eines Transkriptes. Der Unterschied zwischen einer Kopie und einem Transkript besteht darin, dass eine Kopie dem Original gleicht, während ein Transkript aus einem anderen Material, in einer anderen Sprache, mit einem anderen Alphabet oder allgemein in einer anderen Codierung erstellt wird. In der Genetik versteht man darunter die Erstellung eines aus RNA bestehenden Transkriptes im Sinne einer Kopie von einer DNA. Man nennt es Transkript anstatt Kopie, weil das Transkript (die RNA) aus einem etwas
anderen Material als das Original (DNA) besteht. Das der Wikimedia commons entnommene erste Schema zeigt ein Stück entspiralisierte DNA mit getrennten Basenpaaren, wo ein Enzym-Komplex namens DNA-abhängige RNA-Polymerase an einem der beiden DNA-Einzelstränge (dem hier Antisense genannten DNA-Matrizenstrang) einen Transkript genannten und aus RNA bestehenden Gegenstrang produziert. |
|
|
| anonym, The National Human Genome Research Institute, public domain |
| Das zweite Schema soll schrittweise den Vorgang der Transkription darstellen. |

|
| Roland Heynkes, CC BY-SA-4.0 |
 Transkriptionsfaktor nennt man ein Protein, das die Transkription beeinflusst.
Transkriptionsfaktor nennt man ein Protein, das die Transkription beeinflusst.
 Translation ist wie so oft in der Genetik ein ganz simpel aus der englischen Sprache abgeleiteter Fachbegriff, der dementsprechend Übersetzung bedeutet. Blausen Medical Communications zeigt eine schöne Animation der Translation im biologischen Sinne. Übersetzt wird während der Translation die in der DNA codierte Erbinformation in die Reihenfolge der Aminosäuren eines Proteins. Zwischen den Protein-Bauplänen in der DNA und den Ribosomen als Werkzeugen der Translation vermitteln mRNAs als Boten (englisch: messenger), welche Kopien der Gene zu den Ribosomen als Orten der Protein-Synthese transportieren. Ein DNA-Molekül kann hintereinander viele Gene enthalten. Man nennt eine solche Gen-Verpackungseinheit Chromosom. Bei Eukaryonten befinden sich die Chromosomen im Zellkern, aber auch die Mitochondrien besitzen noch ein eigenes Chromosom. In Prokaryoten liegt das Chromosom ohne Hülle in der Zelle. Da es in den Nukleinsäuren DNA und mRNA nur jeweils vier verschiedene Nukleotide, in den Proteinen aber 20 verschiedene Aminosäuren gibt, kann nicht ein einziges Nukleotid für eine Aminosäure stehen. Mit zwei Nukleotiden ließen sich auch nur 42 = 16 verschiedene Aminosäuren codieren. Um 20 verschiedene Aminosäuren codieren zu können, braucht man drei aufeinander folgende Nukleotide, die eine Bedeutungseinheit, also so etwas wie ein Wort bilden. Man nennt diese Worte des genetischen Codes auch Codons, Tripletts, Nukleotid-Tripletts oder Basen-Tripletts, weil sie jeweils 3 Basen enthalten. Mit vier verschiedenen Nukleotiden lassen sich 4 mal 3 = 64 verschiedene Tripletts bilden, aber für die Codierung von nur 20 verschiedenen Aminosäuren würden eigentlich 20 verschiedene Codons ausreichen. Vier Tripletts stehen für den Beginn (Start-Codon AUG) oder das Ende (Stoppcodons UAG, UAA und UGA) der Translation. Das U steht für das Nukleotid Uracil, welches in der mRNA das Desoxythymidin der DNA ersetzt. Während das Startcodon AUG gleichzeitig auch für die Aminosäure Methionin steht, sind die drei (Stoppcodons UAG, UAA und UGA sogenannte Unsinn-Codons, die in keine Aminosäure übersetzt werden. Die 61 codierenden Tripletts stehen für die 20 Aminosäuren, wobei die Aminosäuren von unterschiedlich vielen Codons codiert werden. Während nur das Triplett UGG für die Aminosäure Tryptophan steht, codieren die Tripletts CUA, CUC, CUG, CUT, UUA und UUG die Aminosäure Leucin. Mit Hilfe der sogenannten Codonsonne lässt sich der genetische Code der Tripletts leicht in die jeweiligen Aminosäuren übersetzen. Man muss dazu aber wissen, wo ein Codon anfängt. Verschiebt man nämlich das Leseraster um ein oder zwei Nukleotide, dann ergeben sich völlig andere Tripletts. Als Startpunkt der Übersetzug wählt man daher das Startcodon AUG und liest weiter bis zum ersten auftauchenden Stoppcodon. Das Werkzeug der Übersetzung ist das Ribosom. Ribosomen lesen die in der mRNA enthaltene Erbinformation und vollziehen nach der Anleitung der mRNA die Synthese eines Proteins. Ribosomen schwimmen im Zytoplasma und binden an die Startcodons von mRNA-Molekülen. Gleichzeitig binden auch sogenannte tRNAs mit jeweils drei Nukleotiden an die Codons der mRNA. An jeder tRNA hängt eine Aminosäure, wobei sich die Aminosäure und das aus drei Nukleotiden bestehende Anticodon der tRNA gemäß der Codonsonne entsprechen. Die tRNA verbindet also ganz materiell den genetischen Code mit der jeweils passenden Aminosäure. Das Ribosom muss im Grunde nur noch die nebeneinander liegenden Aminosäuren benachbarter tRNAs miteinander verbinden. Während ein Ribosom vom Kopf zum Schwanz einer mRNA wandert, liest es Codon für Codon und hängt dabei jedesmal eine weitere Aminosäure an die wachsende Peptid-Kette an. Ganz am Anfang der wachsenden Peptid-Kette befindet sich eine Signalsequenz, die den Bestimmungsort des fertigen Proteins verrät. Entsteht da gerade ein später im Zytoplasma gebrauchtes Protein, dann findet die Translation frei schwimmend im Zytoplasma statt. Soll aber das gerade entstehende Protein später aus der Zelle ausgeschleust werden oder auf seiner Zelloberfläche wirken, dann bewirkt die entsprechende Signalsequenz eine Unterbrechung der Translation, bis das Ribosom mit der mRNA zu einem noch freien Platz auf der äußeren Oberfläche des rauhen endoplasmatischen Retikulums gebracht wurde. Dort dockt das Ribosom an und die Peptid-Kette wird durch eine Pore in das endoplasmatische Retikulum eingeführt. Anschließend setzt das Ribosom das Ablesen der mRNA fort und die Peptid-Kette wächst weiter in das endoplasmatische Retikulum hinein. Dort faltet sich die wachsende Peptid-Kette, bis das Protein fertig ist. Im endoplasmatischen Retikulum folgen dann noch sogenannte posttranslationale Modifikationen. Das bedeutet, das noch Kohlenhydrat- und/oder Lipid-Moleküle an die Proteine angehängt werden.
Translation ist wie so oft in der Genetik ein ganz simpel aus der englischen Sprache abgeleiteter Fachbegriff, der dementsprechend Übersetzung bedeutet. Blausen Medical Communications zeigt eine schöne Animation der Translation im biologischen Sinne. Übersetzt wird während der Translation die in der DNA codierte Erbinformation in die Reihenfolge der Aminosäuren eines Proteins. Zwischen den Protein-Bauplänen in der DNA und den Ribosomen als Werkzeugen der Translation vermitteln mRNAs als Boten (englisch: messenger), welche Kopien der Gene zu den Ribosomen als Orten der Protein-Synthese transportieren. Ein DNA-Molekül kann hintereinander viele Gene enthalten. Man nennt eine solche Gen-Verpackungseinheit Chromosom. Bei Eukaryonten befinden sich die Chromosomen im Zellkern, aber auch die Mitochondrien besitzen noch ein eigenes Chromosom. In Prokaryoten liegt das Chromosom ohne Hülle in der Zelle. Da es in den Nukleinsäuren DNA und mRNA nur jeweils vier verschiedene Nukleotide, in den Proteinen aber 20 verschiedene Aminosäuren gibt, kann nicht ein einziges Nukleotid für eine Aminosäure stehen. Mit zwei Nukleotiden ließen sich auch nur 42 = 16 verschiedene Aminosäuren codieren. Um 20 verschiedene Aminosäuren codieren zu können, braucht man drei aufeinander folgende Nukleotide, die eine Bedeutungseinheit, also so etwas wie ein Wort bilden. Man nennt diese Worte des genetischen Codes auch Codons, Tripletts, Nukleotid-Tripletts oder Basen-Tripletts, weil sie jeweils 3 Basen enthalten. Mit vier verschiedenen Nukleotiden lassen sich 4 mal 3 = 64 verschiedene Tripletts bilden, aber für die Codierung von nur 20 verschiedenen Aminosäuren würden eigentlich 20 verschiedene Codons ausreichen. Vier Tripletts stehen für den Beginn (Start-Codon AUG) oder das Ende (Stoppcodons UAG, UAA und UGA) der Translation. Das U steht für das Nukleotid Uracil, welches in der mRNA das Desoxythymidin der DNA ersetzt. Während das Startcodon AUG gleichzeitig auch für die Aminosäure Methionin steht, sind die drei (Stoppcodons UAG, UAA und UGA sogenannte Unsinn-Codons, die in keine Aminosäure übersetzt werden. Die 61 codierenden Tripletts stehen für die 20 Aminosäuren, wobei die Aminosäuren von unterschiedlich vielen Codons codiert werden. Während nur das Triplett UGG für die Aminosäure Tryptophan steht, codieren die Tripletts CUA, CUC, CUG, CUT, UUA und UUG die Aminosäure Leucin. Mit Hilfe der sogenannten Codonsonne lässt sich der genetische Code der Tripletts leicht in die jeweiligen Aminosäuren übersetzen. Man muss dazu aber wissen, wo ein Codon anfängt. Verschiebt man nämlich das Leseraster um ein oder zwei Nukleotide, dann ergeben sich völlig andere Tripletts. Als Startpunkt der Übersetzug wählt man daher das Startcodon AUG und liest weiter bis zum ersten auftauchenden Stoppcodon. Das Werkzeug der Übersetzung ist das Ribosom. Ribosomen lesen die in der mRNA enthaltene Erbinformation und vollziehen nach der Anleitung der mRNA die Synthese eines Proteins. Ribosomen schwimmen im Zytoplasma und binden an die Startcodons von mRNA-Molekülen. Gleichzeitig binden auch sogenannte tRNAs mit jeweils drei Nukleotiden an die Codons der mRNA. An jeder tRNA hängt eine Aminosäure, wobei sich die Aminosäure und das aus drei Nukleotiden bestehende Anticodon der tRNA gemäß der Codonsonne entsprechen. Die tRNA verbindet also ganz materiell den genetischen Code mit der jeweils passenden Aminosäure. Das Ribosom muss im Grunde nur noch die nebeneinander liegenden Aminosäuren benachbarter tRNAs miteinander verbinden. Während ein Ribosom vom Kopf zum Schwanz einer mRNA wandert, liest es Codon für Codon und hängt dabei jedesmal eine weitere Aminosäure an die wachsende Peptid-Kette an. Ganz am Anfang der wachsenden Peptid-Kette befindet sich eine Signalsequenz, die den Bestimmungsort des fertigen Proteins verrät. Entsteht da gerade ein später im Zytoplasma gebrauchtes Protein, dann findet die Translation frei schwimmend im Zytoplasma statt. Soll aber das gerade entstehende Protein später aus der Zelle ausgeschleust werden oder auf seiner Zelloberfläche wirken, dann bewirkt die entsprechende Signalsequenz eine Unterbrechung der Translation, bis das Ribosom mit der mRNA zu einem noch freien Platz auf der äußeren Oberfläche des rauhen endoplasmatischen Retikulums gebracht wurde. Dort dockt das Ribosom an und die Peptid-Kette wird durch eine Pore in das endoplasmatische Retikulum eingeführt. Anschließend setzt das Ribosom das Ablesen der mRNA fort und die Peptid-Kette wächst weiter in das endoplasmatische Retikulum hinein. Dort faltet sich die wachsende Peptid-Kette, bis das Protein fertig ist. Im endoplasmatischen Retikulum folgen dann noch sogenannte posttranslationale Modifikationen. Das bedeutet, das noch Kohlenhydrat- und/oder Lipid-Moleküle an die Proteine angehängt werden.
 Transmembranprotein nennt man ein Protein, das in einer Membran steckt und auf beiden Seiten heraus ragt.
Transmembranprotein nennt man ein Protein, das in einer Membran steckt und auf beiden Seiten heraus ragt.
 transparent = durchsichtig, durchscheinend, Licht durchlassend oder im übertragenen Sinne glaubwürdig, durchschaubar und nachvollziehbar durch Offenlegung aller Informationsquellen, Algorithmen, Parameter, Regeln oder Kriterien
transparent = durchsichtig, durchscheinend, Licht durchlassend oder im übertragenen Sinne glaubwürdig, durchschaubar und nachvollziehbar durch Offenlegung aller Informationsquellen, Algorithmen, Parameter, Regeln oder Kriterien
 Transpiration nennt man das Verdunsten von Wasser durch Pflanzen oder Tiere.
Transpiration nennt man das Verdunsten von Wasser durch Pflanzen oder Tiere.
 Transplantat nennt man einen Körperteil, der von einem Organismus in einen anderen übertragen wird/wurde.
Transplantat nennt man einen Körperteil, der von einem Organismus in einen anderen übertragen wird/wurde.
 Transplantation nennt man die Übertragung oder Verpflanzung eines Körperteils, meistens von einem Organismus auf einen anderen.
Transplantation nennt man die Übertragung oder Verpflanzung eines Körperteils, meistens von einem Organismus auf einen anderen.
 transplantieren = eine Transplantation durchführen
transplantieren = eine Transplantation durchführen
 Transposons sind mobile DNA, die manchmal auch springende Gene genannt werden, obwohl sie gar keine Gene sind. Gemeint ist, dass Transposons neue Positionen im Genom einer Zelle einnehmen und dabei auch dessen Umfang vergrößern oder verkleinern können. Transposons verursachen auch Mutationen, wenn sie in Gene oder in deren Regulationssequenzen hinein springen. Während der Meisose erschweren Sequenzen mit mehreren identischen Sequenzen in Folge die korrekte Paarung und können ungleiche Austausche verursachen.
Transposons sind mobile DNA, die manchmal auch springende Gene genannt werden, obwohl sie gar keine Gene sind. Gemeint ist, dass Transposons neue Positionen im Genom einer Zelle einnehmen und dabei auch dessen Umfang vergrößern oder verkleinern können. Transposons verursachen auch Mutationen, wenn sie in Gene oder in deren Regulationssequenzen hinein springen. Während der Meisose erschweren Sequenzen mit mehreren identischen Sequenzen in Folge die korrekte Paarung und können ungleiche Austausche verursachen.
Es lassen sich drei Klassen (I, II, III) von Transposons unterscheiden:
Klasse-I-Transposons heißen auch Retrotransposons, weil sie zunächst in RNA-Einzelstränge transkribiert und danach durch das Enzym Reverse Transkriptase in DNA-Doppelstränge umgewandelt werden, die schließlich irgendwo als zusätzliche Kopie ins Genom eingebaut werden können. Deshalb werden sie im Genom nicht verschoben, sondern kopiert. Ihre Zahl nimmt daher immer nur zu und insgesamt sollen sie inzwischen bis zu 40% des menschlichen Genoms ausmachen. Viele Retrotransposons besitzen an ihren Enden sogenannte long terminal repeats (LTR), die über 1000 Nukleotide lang sein können. Genau wie bei Klasse-II-Transposons führt auch auch der Einbau eines Retrotransposons an der Insertionsstelle beiderseits des Transposons zu kurzen direct repeats (identischen Sequenzen auf beiden Seiten).
Long interspersed elements (LINE) nennt man Retrotransposons, von denen das menschliche Genom rund 850.000 LINEs enthalten soll, die insgesamt etwa 21% unseres Genoms ausmachen sollen. Die meisten davon gehören zur Familie der LINE-1 (L1), die wenige Hundert bis 9.000 Nukleotide lang sein können. Nur etwa 50 L1-Elemente enthalten funktionelle "Gene" die transkribiert und translatiert werden. Diese funktionellen L1-Elemente sind rund 6.500 Basenpaare lang und kodieren 3 Proteine. Die RNA-Polymerase II transkribiert die L1-DNA in RNA, die dann teilweise von zellulären Ribosomen in die 3 Proteine translatiert wird. Aus dem Zytoplasma gelangen die RNA und die Proteine in den Zellkern zurück. Hier macht eine Reverse Transkriptase aus dem RNA-Transkript eine doppelsträngige DNA-Kopie. Die Endonuklease schneidet die DNA am Zielort auf, sodaß die Kopie des L1-Elementes dort eingefügt werden kann. Viel schneller als normale Mutationen sorgt so die unterschiedliche Verteilung der LINEs im Genom für individuelle Unterschiede und generiert damit genetische Fingerabdrücke. Die Variabilität wird noch dadurch gesteigert, daß die Transkription der L1-Elemente gelegentlich am 3'-Ende über das Transposon hinaus geht. Die Kopie des Transposons wird dadurch größer. Andererseits bricht die DNA-Synthese durch die Reverse Transkriptase häufig vorzeitig ab, wodurch verkürzte Transposon-Versionen entstehen. Werden LINE-Kopien in regulierende Regionen integriert oder verlängern sie als Bestandteil eines Introns deutlich die Länge des primären Transkripts, dann können sie Einfluß auf die Expression einzelner Gene nehmen. Manchmal wird mit einem LINE auch eine kodierende Gen-Sequenz kopiert. Dann kann sich aus dieser Kopie ein neues Gen entwickeln. Zunächst entstehen so aber nur Pseudogene ohne Introns und Kontrollsequenzen.
Short interspersed elements (SINE) sind nur 100-400 Nukleotide lang und enthalten von Reverser Transkriptase synthetisierte Sequenzen aus tRNA, 5S rRNA und andere kleine Kern-RNAs, die von der RNA-Polymerase III transkribiert werden. Der häufigste SINE-Typ ist das vermutlich aus 7S RNA entstandene 300 Nukleotide lange und eine namensgebende Erkennungssequenz der Restriktionsendonuklease AluI enthaltende Alu-Element, von welchem das menschliche Genom über 1 Million Kopien bzw. 11% des Gesamtgenoms enthält. Die meisten SINE kodieren keine funktioniellen Peptide oder RNA und werden passiv von den Enzymen mitkopiert, die von aktiven L1-Elementen kodiert werden. SINE verursachen relativ wenige Mutationen.
Retroviren wie HIV funktionieren innerhalb des Zellkerns auch wie Retrotransposons.
Klasse-II-Transposons sind direkt von einer Position im Genom zu einer anderen wechselnde DNA-Bereiche. Mit Hilfe einer Transposase werden sie enzymatisch aus ihrer Umgebung herausgeschnitten und an anderer Stelle wieder eingefügt. Einige Klasse-II-Transposons kodieren selber dieses Enzym, welches beide Enden eines Transposons sowie eine Zielsequenz an einer anderen Stelle in der DNA bindet. Einige Transposasen benötigen dafür spezifische Erkennungssequenzen, während andere ihr Transposon an jeder beliebigen Stelle integrieren können. Die Zielsequenz wird so geschnitten, das einer der beiden DNA-Stänge um einige Nukleotide übersteht und ein sogenanntes sticky end entsteht. Das Transposon selber hat aber keine sticky ends, sondern sogenannte blunt ends, sodaß nach dem Einfügen (Ligation) die beiden kurzen nur einzelsträngigen Bereiche noch mit einigen Nukleotiden aufgefüllt werden müssen. Dadurch entstehen direkt außerhalb der inverted repeats des Transposons noch zwei identische direct repeats.
Vor etwa 50 Jahren entstand erstmals in der Fruchtfliege Drosophila ein Klasse-II-Transposon, das P-Element genannt wird. Normalerweise wird dessen Transposase-Gen nicht exprimiert. Wenn allerdings eine männliche Fliege mit diesem Element Nachkommen mit einer weiblichen Fliege ohne es bekommt, dann wird in den Nachkommen das Transposase-Gen des P-Elementes exprimiert. Dadurch entstehen auch in der Keimbahn so viele Mutationen, daß die Nachkommen steril werden. Trotzdem hat sich dieses Klasse-II-Transposon in der Fruchtfliege so schnell vermehrt, daß man Drosophila ohne es nur noch in alten Zuchtstämmen, aber nicht mehr in der Natur findet. Molekularbiologen konnten das P-Element als Genfähre nutzen. Klasse-III-Transposons nennt man auch Miniature Inverted-repeats Transposable Elements oder MITE. Im Genom von Reis wurden mehr als 100.000 MITEs gefunden. Bei einer Länge von jeweils etwa 400 Basenpaaren und ihren flankierenden jeweils rund 15 Basenpaare langen inverted repeats repräsentieren sie insgesamt etwa 6% des Reisgenoms. Sie sind zu klein, um ein Enzym zu kodieren, und man weiß noch nicht, wie sie sich so ausbreiten konnten. Vielleicht wurden sie von der Transposase eines größeren Klasse-II-Transposons kopiert, welches die selben inverted repeats besitzt. MITEs wurden auch in den Genomen von Mensch, Xenopus und Apfel gefunden.
Einige bakterielle Transposons transportieren auch in der Natur neben dem Transposase-Gen mindestens ein Antibiotikumresistenzgen. Natürlich springen solche Transposons auch in Plasmide, mit deren Hilfe sie dann auf andere Bakterien übertragen werden können. Außer dem Transposase-Gen besitzen diese Transposons auch ein Gen für das Enzym Resolvase, welches dafür sorgt, daß das Transposon nicht ausgeschnitten, sonden kopiert wird.
 Traube nennen Botaniker einen unverzweigten Blütenstand, bei dem die gestielten Blüten nicht am Ende, sondern alle nur seitlich und von der Sprossachse ausgehen. Allerdings wissen das längst nicht alle Biologen. Wie die meisten Deutschen meinen auch die meisten Biologen meistens Weintrauben, wenn sie von Trauben sprechen oder schreiben. Nur die Botaniker glauben es besser zu wissen und erklären, Weintrauben seien gar keine Trauben, sondern entwickelten sich aus Rispen.
Traube nennen Botaniker einen unverzweigten Blütenstand, bei dem die gestielten Blüten nicht am Ende, sondern alle nur seitlich und von der Sprossachse ausgehen. Allerdings wissen das längst nicht alle Biologen. Wie die meisten Deutschen meinen auch die meisten Biologen meistens Weintrauben, wenn sie von Trauben sprechen oder schreiben. Nur die Botaniker glauben es besser zu wissen und erklären, Weintrauben seien gar keine Trauben, sondern entwickelten sich aus Rispen.
 Trauma nennt man eine körperliche oder seelische Verletzung, die durch eine starke äußere Einwirkung verursacht wurde.
Trauma nennt man eine körperliche oder seelische Verletzung, die durch eine starke äußere Einwirkung verursacht wurde.
 traumatisch = Traumatisierung verursachend, ein Trauma betreffend oder durch ein Trauma entstanden
traumatisch = Traumatisierung verursachend, ein Trauma betreffend oder durch ein Trauma entstanden
 Traumatisierung nennt man die Entstehung einer dauerhaften seelischen Verletzung aufgrund eines schrecklichen Erlebnisses.
Traumatisierung nennt man die Entstehung einer dauerhaften seelischen Verletzung aufgrund eines schrecklichen Erlebnisses.
 Treibhauseffekt nennt man die Erwärmung eines Planeten durch eine Atmosphäre, in der sogenannte Treibhausgase ähnlich wie die Fenster eines Treibhauses einen Teil der vom Planeten abgestrahlten Wärmestrahlung am Entweichen in das Universum hindern.
Treibhauseffekt nennt man die Erwärmung eines Planeten durch eine Atmosphäre, in der sogenannte Treibhausgase ähnlich wie die Fenster eines Treibhauses einen Teil der vom Planeten abgestrahlten Wärmestrahlung am Entweichen in das Universum hindern.
 Tremor = Zittern, verursacht durch anhaltende rhythmische Kontraktionen einzelner Muskeln oder Muskelgruppen (Muskeltremor), auch Zitterbewegungen (des Kopfes oder der Gliedmaßen) (Dr. Ingrid Schütt-Abraham)
Tremor = Zittern, verursacht durch anhaltende rhythmische Kontraktionen einzelner Muskeln oder Muskelgruppen (Muskeltremor), auch Zitterbewegungen (des Kopfes oder der Gliedmaßen) (Dr. Ingrid Schütt-Abraham)
 Trias heißt das Erdzeitalter, das vor 252 Millionen Jahren nach dem Ende des Erdzeitalters Paläozoikum begann und vor 201 Millionen Jahren mit dem Beginn des Erdzeitalters Jura endete.
Trias heißt das Erdzeitalter, das vor 252 Millionen Jahren nach dem Ende des Erdzeitalters Paläozoikum begann und vor 201 Millionen Jahren mit dem Beginn des Erdzeitalters Jura endete.
 Trigeminus = Nervus trigeminus
Trigeminus = Nervus trigeminus
 Trigeminusganglien = Das Ganglion trigeminale oder Ganglion Gasseri liegt in einer Impressio trigemini genannten Mulde auf der das Innenohr umgebenden Felsenbeinpyramide
Trigeminusganglien = Das Ganglion trigeminale oder Ganglion Gasseri liegt in einer Impressio trigemini genannten Mulde auf der das Innenohr umgebenden Felsenbeinpyramide
 Trillion ist ein Zahlwort, dass im Deutschen in Frankreich einer Million hoch 3 (daher die Vorsilbe tri) entspricht, also 1018, eine Eins mit 18 Nullen. In einem Biologie-Glossar müsste man das nicht erwähnen, würden nicht ständig amerikanische und englische Dokumentationen falsch übersetzt. Seltsamerweise wissen die meisten Übersetzer wissenschaftlicher Fernsehsendungen nicht, dass Amerikaner und manche Briten 1012 meinen, wenn sie von einer Trillion sprechen. Ist also in einer ins Deutsche übersetzten US-amerikanischen oder britischen Sendung von einer Trillion die Rede, dann sollte man immer überprüfen, ob es nicht im Deutschen Billion heißen müsste. Eine US-amerikanische Billion entspricht einer deutschen Milliarde.
Trillion ist ein Zahlwort, dass im Deutschen in Frankreich einer Million hoch 3 (daher die Vorsilbe tri) entspricht, also 1018, eine Eins mit 18 Nullen. In einem Biologie-Glossar müsste man das nicht erwähnen, würden nicht ständig amerikanische und englische Dokumentationen falsch übersetzt. Seltsamerweise wissen die meisten Übersetzer wissenschaftlicher Fernsehsendungen nicht, dass Amerikaner und manche Briten 1012 meinen, wenn sie von einer Trillion sprechen. Ist also in einer ins Deutsche übersetzten US-amerikanischen oder britischen Sendung von einer Trillion die Rede, dann sollte man immer überprüfen, ob es nicht im Deutschen Billion heißen müsste. Eine US-amerikanische Billion entspricht einer deutschen Milliarde.
 Triplett ist ein in der Genetik übliches Kürzel für Basentriplett.
Triplett ist ein in der Genetik übliches Kürzel für Basentriplett.
 Tris-HCl = Tris(hydroxymethyl)-aminomethanhydrochlorid (C4H11NO3ClH)
Tris-HCl = Tris(hydroxymethyl)-aminomethanhydrochlorid (C4H11NO3ClH)
 Tricuspidalklappe = dreizipflige, von Muskeln gehaltene Segelklappe, die den rechten Herzvorhof von der rechten Herzkammer trennt und beim Ausstoßen des Blutes aus der rechten Herzkammer in die Lungenarterien verhindert, dass das Blut in den rechten Vorhof zurückfließt (Dr. Ingrid Schütt-Abraham)
Tricuspidalklappe = dreizipflige, von Muskeln gehaltene Segelklappe, die den rechten Herzvorhof von der rechten Herzkammer trennt und beim Ausstoßen des Blutes aus der rechten Herzkammer in die Lungenarterien verhindert, dass das Blut in den rechten Vorhof zurückfließt (Dr. Ingrid Schütt-Abraham)
 Trimmen = Entfernen oberflächlich kontaminierter, veränderter oder unerwünschter Teile des Schlachtkörpers, regional auch "Putzen" genannt (Dr. Ingrid Schütt-Abraham)
Trimmen = Entfernen oberflächlich kontaminierter, veränderter oder unerwünschter Teile des Schlachtkörpers, regional auch "Putzen" genannt (Dr. Ingrid Schütt-Abraham)
 trivial = einfach, normal, unbedeutend, einfallslos, nichtssagend, banal
trivial = einfach, normal, unbedeutend, einfallslos, nichtssagend, banal
 Trivialnamen nennt man in Chemie und Biochemie Namen, die sich im Laufe der Zeit ohne einheitliche Systematik eingebürgert haben.
Trivialnamen nennt man in Chemie und Biochemie Namen, die sich im Laufe der Zeit ohne einheitliche Systematik eingebürgert haben.
 Trizeps (mit drei Köpfen bzw. Ansatzpunkten) oder Strecker (nach seiner Funktion) heißt der Muskel, der das Ellbogen-Gelenk strecktt. Der Trizeps ist der Antagonist des Bizeps.
Trizeps (mit drei Köpfen bzw. Ansatzpunkten) oder Strecker (nach seiner Funktion) heißt der Muskel, der das Ellbogen-Gelenk strecktt. Der Trizeps ist der Antagonist des Bizeps.
| tRNA ist die englischsprachige Abkürzung für transfer-RNA. Diese etwa 80 Nukleotide langen und durch intramolekulare Basenpaarungen winkelförmig gefaltete Ribonukleinsäuren besitzen eine aus drei benachbarten Nukleotiden (Triplett) bestehende RNA-Erkennungssequenz, mit der sie an den der Protein-Biosynthese dienenden Ribosomen jeweils an ein zur Erkennungssequenz komplementäres Codon der Boten-RNA binden. Außer dem auch Anticodon genannten Triplett besitzt jede tRNA noch eine Bindungsstelle für eine Aminosäure. |
 |
| Das Enzym Aminoacyl-tRNA Synthetase belädt jede tRNA in Abhängigkeit von ihrer RNA-Erkennungssequenz mit der Aminosäure, die dem Codon entspricht, welches von der RNA-Erkennungssequenz der tRNA erkannt wird. Wenn nun an einem Ribosom die nebeneinander liegenden Codons der RNA die jeweils zu ihnen passenden tRNAs gebunden haben, dann liegen auch die an den tRNAs hängenden Aminosäuren nebeneinander und werden am Ribosom in der von der RNA vorgegebenen Reihenfolge miteinander zu einem neuen Protein verknüpft. |
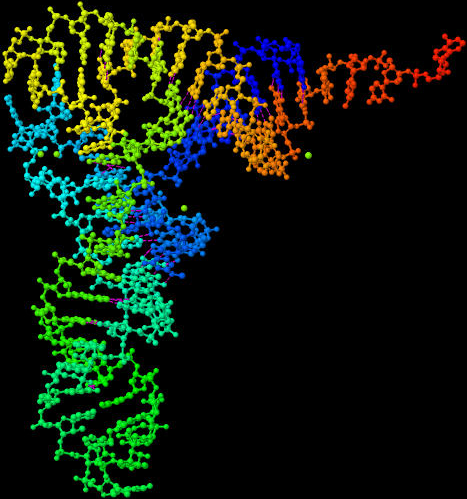 |
| Die drei Bilder zeigen die Hefe-tRNA für die Aminosäure Phenylalanin. Gestrichelt sind die Wasserstoffbrückenbindungen angedeutet. Erstellt habe ich die Bilder mit dem Java-Programm Jmol und den Koordinaten der Sequenz 4TNA der frei zugänglichen Protein Data Bank und dem Jmol-applet des Open Source Java Viewer for chemical structures in 3D. Diese Ergebnisse meiner Nutzung frei verfügbarer Daten und Programme dürfen selbstverständlich ebenfalls von jedem völlig frei genutzt werden. |
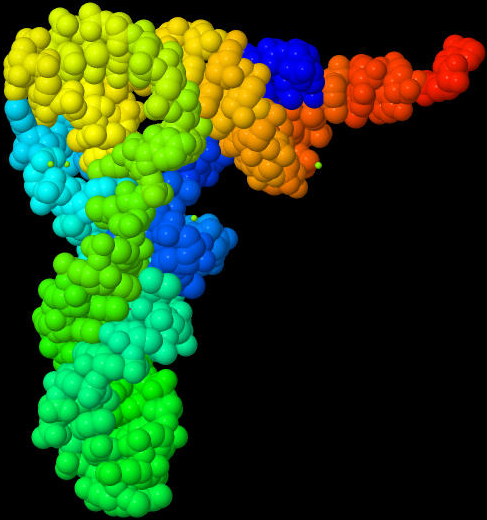 |
 Trochanter nennt man bei Säugetieren 2-3 als Ansatzflächen verschiedener Muskeln dienende Vorsprünge am Oberschenkelknochen. Bei Gliederfüßern nennt man das zweite Glied des Beins (zwischen Coxa und Femur) Trochanter oder Schenkelring.
Trochanter nennt man bei Säugetieren 2-3 als Ansatzflächen verschiedener Muskeln dienende Vorsprünge am Oberschenkelknochen. Bei Gliederfüßern nennt man das zweite Glied des Beins (zwischen Coxa und Femur) Trochanter oder Schenkelring.
 Trogocytose oder Trogozytose heißt eine Endozytose zwischen Pinozytose und Phagozytose. Pino kommt von Trinken, Phago von Fressen und Trogo von Knabbern. Trogozytose heißt also eine Endozytose, bei der eine Zelle kleine Stücke aus der Zellmembran einer anderen Zelle heraus knabbert.
Trogocytose oder Trogozytose heißt eine Endozytose zwischen Pinozytose und Phagozytose. Pino kommt von Trinken, Phago von Fressen und Trogo von Knabbern. Trogozytose heißt also eine Endozytose, bei der eine Zelle kleine Stücke aus der Zellmembran einer anderen Zelle heraus knabbert.
Entdeckt wurde dieser Mechanismus zuerst in den späten 1970er Jahren bei Lymphozyten (B-, T- und NK-Zellen B-Lymphozyten, T-Lymphozyten und natürlichen Killerzellen), die auf diese Weise Antigene von Antigen-präsentierenden Zellen übernehmen.
| Trogocytose bei Lymphozyten |
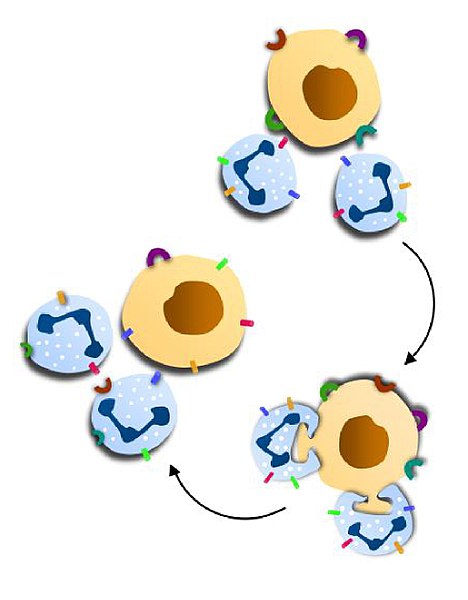 |
| Dana Weidner, CC-by-sa 2.0/de |
 Tropen heißen beiderseits des Äquators die ständig heißen Gebiete zwischen den Wendekreisen bei 23,5° nördlicher bzw. südlicher Breite. In den Tropen steht die Sonne mindestens einmal im Jahr im Zenit und die Tageslängen schwanken im Jahresverlauf nur relativ gering zwischen 10,5 und 13,5 Stunden.
Tropen heißen beiderseits des Äquators die ständig heißen Gebiete zwischen den Wendekreisen bei 23,5° nördlicher bzw. südlicher Breite. In den Tropen steht die Sonne mindestens einmal im Jahr im Zenit und die Tageslängen schwanken im Jahresverlauf nur relativ gering zwischen 10,5 und 13,5 Stunden.
 trophisches Hormon, Freisetzungshormon, Releasing-Hormon oder Liberin nennt man ein Hormon, welches die Bildung anderer Hormone anregt.
trophisches Hormon, Freisetzungshormon, Releasing-Hormon oder Liberin nennt man ein Hormon, welches die Bildung anderer Hormone anregt.
 Trophoblast heißt die äußere Zellschicht einer Blastozyste und verbindet diese mit der Gebärmutterwand. Der Trophoblast bildet sich am 5. bis 12. Tag nach der Befruchtung aus Blastomeren, weicht mittels Enzymen die Gebärmutter-Schleimhaut auf und kann sich so an ihr festsetzen. Trophoblasten haben keine MHC-1-Präsentierteller.
Trophoblast heißt die äußere Zellschicht einer Blastozyste und verbindet diese mit der Gebärmutterwand. Der Trophoblast bildet sich am 5. bis 12. Tag nach der Befruchtung aus Blastomeren, weicht mittels Enzymen die Gebärmutter-Schleimhaut auf und kann sich so an ihr festsetzen. Trophoblasten haben keine MHC-1-Präsentierteller.
 Trophiestufe nennt man eine Stufe in einer Nahrungskette.
Trophiestufe nennt man eine Stufe in einer Nahrungskette.
 tropisch = die Tropen betreffend, in den Tropen, typisch für die Tropen
tropisch = die Tropen betreffend, in den Tropen, typisch für die Tropen
 Tropischer Regenwald oder genauer tropischer Tiefland-Regenwald nennt man die Wälder rund um den Äquator, auf die sehr viel Regen fällt. Die Wolken entstehen zu einem erheblichen Teil durch die Verdunstung von Wasser durch die Blätter der Bäume.
Tropischer Regenwald oder genauer tropischer Tiefland-Regenwald nennt man die Wälder rund um den Äquator, auf die sehr viel Regen fällt. Die Wolken entstehen zu einem erheblichen Teil durch die Verdunstung von Wasser durch die Blätter der Bäume.
 Truncus encephali, Truncus cerebri oder Hirnstamm ist wie das Großhirn ein nicht wirklich klar definierter Begriff der makroskopischen Humananatomie für den unpaarigen Teil des menschlichen Gehirns, welcher das Rückenmark gradlinig fortsetzt und aus dem sich Großhirn und Kleinhirn jeweils paarig mit ihren Hemisphären ausstülpen. Im Hirnstamm entspringen die motorischen und enden die sensorischen Anteile der meisten Hirnnerven. Eindeutig zum Hirnstamm gehören das Mittelhirn (Mesencephalon) sowie das aus Hinterhirn (Metencephalon) und Nachhirn (Myelencephalon) bestehende Rautenhirn (Rhombencephalon) und der Pschyrembel belässt es dabei. Laut Brockhaus umfasst es aber außerdem das Zwischenhirn (Diencephalon) sowie die Basalganglien des Endhirns (Telencephalon), was einer Überschneidung von Hirnstamm und Endhirn (Telencephalon) gleich käme. Auch das Duden-Wörterbuch medizinischer Fachausdrücke zählt zum Hirnstamm das Großhirn mit Ausnahme des Hirnmantels (Pallidum). Da mag sich das Online-Lexikon der Neurowissenschaft gar nicht erst festlegen und stellt einfach nur die Konfusion fest. Mehr kann ich auch nicht tun, obwohl eine Überschneidung von Großhirn (Cerebrum), Kleinhirn (Cerebellum) und Hirnstamm nicht sinnvoll wirkt.
Truncus encephali, Truncus cerebri oder Hirnstamm ist wie das Großhirn ein nicht wirklich klar definierter Begriff der makroskopischen Humananatomie für den unpaarigen Teil des menschlichen Gehirns, welcher das Rückenmark gradlinig fortsetzt und aus dem sich Großhirn und Kleinhirn jeweils paarig mit ihren Hemisphären ausstülpen. Im Hirnstamm entspringen die motorischen und enden die sensorischen Anteile der meisten Hirnnerven. Eindeutig zum Hirnstamm gehören das Mittelhirn (Mesencephalon) sowie das aus Hinterhirn (Metencephalon) und Nachhirn (Myelencephalon) bestehende Rautenhirn (Rhombencephalon) und der Pschyrembel belässt es dabei. Laut Brockhaus umfasst es aber außerdem das Zwischenhirn (Diencephalon) sowie die Basalganglien des Endhirns (Telencephalon), was einer Überschneidung von Hirnstamm und Endhirn (Telencephalon) gleich käme. Auch das Duden-Wörterbuch medizinischer Fachausdrücke zählt zum Hirnstamm das Großhirn mit Ausnahme des Hirnmantels (Pallidum). Da mag sich das Online-Lexikon der Neurowissenschaft gar nicht erst festlegen und stellt einfach nur die Konfusion fest. Mehr kann ich auch nicht tun, obwohl eine Überschneidung von Großhirn (Cerebrum), Kleinhirn (Cerebellum) und Hirnstamm nicht sinnvoll wirkt.
Hirnstamm in einem rotierenden Modell des menschlichen Hirns 
|
|---|
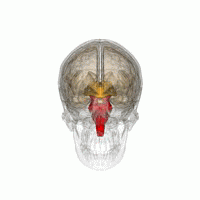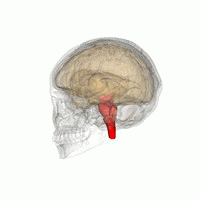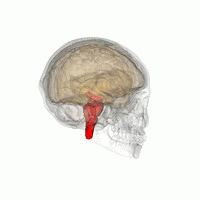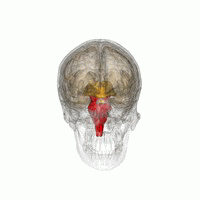 Für eine größere Version klicke auf das rotierende Bild.
Für eine größere Version klicke auf das rotierende Bild.
|
| BodyParts3D/Anatomography, The Database Center for Life Science, CC-BY-SA-2.1-jp |
| Dieses rotierende Modell eines menschlichen Hirns hebt den Hirnstamm rot hervor. |
 Truncus vagosympathicus = aus sympathischen und parasympathischen Anteilen bestehender und im Halsbereich gelegener Abschnitt des Nervus vagus (Dr. Ingrid Schütt-Abraham)
Truncus vagosympathicus = aus sympathischen und parasympathischen Anteilen bestehender und im Halsbereich gelegener Abschnitt des Nervus vagus (Dr. Ingrid Schütt-Abraham)
 TSE = Transmissible Spongiform Encephalopathy oder übertragbare schwammartige Hirndegeneration, von Anhängern der Protein-only-Hypothese auch Prionkrankheiten genannt
TSE = Transmissible Spongiform Encephalopathy oder übertragbare schwammartige Hirndegeneration, von Anhängern der Protein-only-Hypothese auch Prionkrankheiten genannt
 Tubuli = röhrenförmige Auswüchse
Tubuli = röhrenförmige Auswüchse
 Tuff heißt ein sehr poröses und deshalb sehr leichtes und relativ weiches Gestein, das entsteht, wenn die Mischung aus heißen Gasen und Asche einer pyroklastischen Wolke erkältet und fest wird.
Tuff heißt ein sehr poröses und deshalb sehr leichtes und relativ weiches Gestein, das entsteht, wenn die Mischung aus heißen Gasen und Asche einer pyroklastischen Wolke erkältet und fest wird.
 Tumor (lateinisch für Wucherung, Geschwulst oder Schwellung) heit im weiteren medizinischen Sinne einfach jede Zunahme des Volumens eines Gewebes. Normalerweise wird der Begriff Tumor aber auch von Medizinern nur im engeren Sinne gebraucht. Gemeint ist dann eine Wucherung aufgrund der übermäßigen Vermehrung der Nachkommen einer Zelle, in der eine Mutation oder eine epigenetische Fehlsteuerung die normale Regulation der Zellteilung außer Kraft gesetzt hat. Wenn in den Tumorzellen sonst nichts außer Kontrolle geraten ist, spricht man von einem gutartigen Tumor, weil er nicht in das ihn umgebende gesunde Gewebe hinein wächst, sondern dieses "nur" zur Seite schiebt. In gutartigen Tumoren kommt es oft zu weiteren Mutationen und manche machen aus einem gutartigen einen bösartigen Tumor, der in die umgebenden Gewebe hinein wächst und diese zerstört. Es geht aber noch schlimmer, wenn eine weitere Mutation dazu führt, dass ein Tumor Metastasen bildet, indem einige seiner Zellen sich ablösen, mit dem Blut in andere Teile des Organismus gelangen und dort neue bösartige Tumore bilden.
Tumor (lateinisch für Wucherung, Geschwulst oder Schwellung) heit im weiteren medizinischen Sinne einfach jede Zunahme des Volumens eines Gewebes. Normalerweise wird der Begriff Tumor aber auch von Medizinern nur im engeren Sinne gebraucht. Gemeint ist dann eine Wucherung aufgrund der übermäßigen Vermehrung der Nachkommen einer Zelle, in der eine Mutation oder eine epigenetische Fehlsteuerung die normale Regulation der Zellteilung außer Kraft gesetzt hat. Wenn in den Tumorzellen sonst nichts außer Kontrolle geraten ist, spricht man von einem gutartigen Tumor, weil er nicht in das ihn umgebende gesunde Gewebe hinein wächst, sondern dieses "nur" zur Seite schiebt. In gutartigen Tumoren kommt es oft zu weiteren Mutationen und manche machen aus einem gutartigen einen bösartigen Tumor, der in die umgebenden Gewebe hinein wächst und diese zerstört. Es geht aber noch schlimmer, wenn eine weitere Mutation dazu führt, dass ein Tumor Metastasen bildet, indem einige seiner Zellen sich ablösen, mit dem Blut in andere Teile des Organismus gelangen und dort neue bösartige Tumore bilden.
 Tumorgewebe heißt das Gewebe, aus dem ein Tumor besteht. Allerdings sind die Tumorzellen in einem Tumor nicht alle gleich, sondern man unterscheidet sich langsam teilende und dabei nicht differenzierende Tumor-Stammzellen von nicht mehr unendlich oft teilungsfähigen normalen Tumorzellen.
Tumorgewebe heißt das Gewebe, aus dem ein Tumor besteht. Allerdings sind die Tumorzellen in einem Tumor nicht alle gleich, sondern man unterscheidet sich langsam teilende und dabei nicht differenzierende Tumor-Stammzellen von nicht mehr unendlich oft teilungsfähigen normalen Tumorzellen.
 Tumorzelle = Zelle n bzw. aus einem Tumor
Tumorzelle = Zelle n bzw. aus einem Tumor
 Tümpel nennt man ein kleines, flaches, stehendes Gewässer, dass auch austrocknen kann.
Tümpel nennt man ein kleines, flaches, stehendes Gewässer, dass auch austrocknen kann.
 Tundra nennt man ein Gebiet, in dem es so kalt ist, das zwar Flechten und Pflanzen, aber keine Bäume wachsen können.
Tundra nennt man ein Gebiet, in dem es so kalt ist, das zwar Flechten und Pflanzen, aber keine Bäume wachsen können.
 Tunica conjunctiva oder Bindehaut heißt eine am Rand der Augenhornhaut beginnende, auf dem sichtbaren Teil der Lederhaut locker aufliegende und fest verwachsen die Rückseiten der Augenlidern bedeckende, gut durchblutete und empfindliche Nervenenden besitzende Schleimhaut.
Tunica conjunctiva oder Bindehaut heißt eine am Rand der Augenhornhaut beginnende, auf dem sichtbaren Teil der Lederhaut locker aufliegende und fest verwachsen die Rückseiten der Augenlidern bedeckende, gut durchblutete und empfindliche Nervenenden besitzende Schleimhaut.
 Typisierung = Charakterisierung einer Variante durch Ermittlung eines Profils ihrer Eigenschaften
Typisierung = Charakterisierung einer Variante durch Ermittlung eines Profils ihrer Eigenschaften